Resumen
Introducción: los cultivos celulares se utilizan frecuentemente en la investigación biomédica, en el área industrial y en pruebas diagnósticas en los hospitales. La contaminación de los cultivos celulares por microorganismos como los micoplasmas o por contaminación cruzada entre cultivos es uno de los principales problemas en la investigación.
Objetivo: detección de micoplasmas en cultivos celulares provenientes de diferentes laboratorios biomédicos.
Material y métodos: los cultivos celulares se evaluaron para detectar presencia de micoplasmas por medio de cultivo microbiológico y PCR. Los cultivos celulares fueron crecidos en ausencia de antibióticos durante 3-4 días. La identificación se presentó por el cambio en el indicador de pH y ausencia de turbidez en el caldo y por la presencia en agar de las colonias características a través de microscopia estereoscópica. La comparación de los resultados obtenidos entre el método microbiológico y PCR se realizó con la prueba de T de Student.
Resultados: por el método microbiológico se detectó en 9/20 muestras (45%) presencia de micoplasmas en los cultivos celulares y por medio de la prueba de PCR resultaron positivas 10/20 muestras (50%) de cultivos celulares. El análisis estadístico mostró que no hay diferencia significativa (P>0.05) en la detección entre el método microbiológico y PCR.
Conclusiones: es recomendable que la detección de micoplasmas en cultivos celulares debe reforzarse con otra técnica para validar los resultados. Rev.cienc.biomed.2010; 1 (2): 187 – 189
Palabras claves: Cultivos celulares. Contaminación. Micoplasmas. Detección.
Rivera Tapia José Antonio1
Castillo Viveros Linda Valeria2
Sánchez Hernández José Antonio3
Correspondencia: jart70@yahoo.com
Recibido para revisión: junio 12 2010
Aceptado para publicación: octubre 10 2010
Summary
Introduction: cells cultures are widely used in both biomedical and biotechnological research centers and industry, as well as for diagnostic test in hospitals. Contaminations of cells cultures with microbial organisms as well as with virus or other eukaryotic cell
Laboratorio de Micoplasmas. Centro de Investigaciones Microbiológicas del Instituto de Ciencias de la Benemérita. Universidad Autónoma de Puebla (BUAP). 2 Escuela de Biología de la BUAP. 3 Laboratorio de Biología Celular, Facultad de Medicina de la BUAP lines are a major problem in cell culture related research.
Objective: mycoplasmas detection in cells cultures came from biomedical laboratories.
Material and methods: the cells cultures screened for mycoplasmas by using of microbiological culture and PCR. Cells cultures were grown in the absence of antibiotics for 3-4 days. The identified based on alterations in the pH of the broth in the absence of turbidity, production of fried egg colonies. Comparison between microbiological and PCR were made using Student T test.
Results: mycoplasmas were detected by culture in 9/20 (45%) of the cell culture samples and PCR revealed the presence of target DNA in 10/20 (50%) samples. Mycoplasmas detection between the microbiological culture and PCR showing not significant differences (P>0.05).
Conclusion: mycoplasmas detection in cell cultures must be strengthen with another technique to validate the results.
Keywords: Cells cultures. Contamination. Mycoplasmas. Detection
Introducción
Los micoplasmas son los microorganismos más pequeños y autorreplicables, se caracterizan por presentar un genoma reducido en comparación con otros procariontes, también carecen de pared celular lo cual condiciona que presenten formas pleomórficas.
Su distribución es considerable ya que los podemos encontrar en diversos animales y en el humano y se reporta que pueden interferir en la investigación biomédica ya que infectan animales de laboratorio o contaminando cultivos celulares (1, 4).
La contaminación en cultivos celulares por micoplasmas puede inducir efectos citogenéticos, disminución en las concentraciones de nutrientes, alteración de la morfología celular, modulación de la respuesta inmune e incluso interrupción del metabolismo celular (5,6).
Si la contaminación se vuelve persistente se dificulta la detección y el diagnóstico, siendo difícil eliminar la contaminación en los cultivos celulares y la concentración de micoplasmas puede llegar hasta 107 UFC/ml (7). La identificación de micoplasmas implica ciertas dificultades ya que su metabolismo no produce turbidez en el medio, estos microorganismos no se tiñen por la técnica de gram y su crecimiento colonial en medio sólido además de ser lento se requiere de observar al microscopio.
De tal forma se recomienda para la detección de micoplasmas en cultivos celulares implementar al menos dos pruebas, ya que se ha visto que se pueden generar resultados falsos positivos o falsos negativos. El objetivo del presente trabajo fue la detección de micoplasmas en cultivos celulares provenientes de diferentes laboratorios biomédicos.
Materiales y métodos para la detección de micoplasmas en cultivos celulares
Del periodo de octubre 2008 a diciembre 2009 se determinó la presencia de micoplasmas en veinte muestras de cultivos celulares. Y en veinte muestras de medios utilizados para crecer los cultivos celulares. El total de muestras se obtuvo de diferentes laboratorios biomédicos de la Ciudad de México, D.F. La detección de micoplasmas se realizó por método microbiológico, con medios específicos para micoplasmas y por medio de la técnica de PCR. (Ver: Cáncer de apéndice)
Cultivos celulares sin antiobióticos
Los cultivos celulares fueron crecidos en ausencia de antibióticos:
Durante 3 – 4 días, enseguida se tomaron alícuotas de 1 ml, cada alícuota se adicionó a 1 ml de caldo SP4 e incubaron a 37ºC durante veinte días. Cada tercer día se resembraron 5 μl en agar SP4 e incubaron en las mismas condiciones. La identificación se presentó por el cambio en el indicador de pH y ausencia de turbidez en el caldo y por la presencia en agar de las colonias características a través de microscopia estereoscópica. Los medios utilizados para crecer los cultivos celulares recibieron el mismo esquema de trabajo.
Para la detección por PCR se tomaron alícuotas de 1 ml de los cultivos y se realizó la extracción de ADN con ZR Fungal/Bacterial DNA kit™ (Zymo Research). Los Primers AR1 y AR2 que amplifican un fragmento de 301 pares de bases en 30 especies de micoplasmas fueron utilizados en el presente trabajo.
Para realizar la amplificación se utilizaron 10μl de PyroStart™ fast PCR Master Mix (2X), 1 μl de cada uno de los primers, 5μl de ADN problema y 3 μl de agua libre de nucleasas, para un volumen total de 20 μl. A continuación se realizó la amplificación en un termociclador (Techne TC-412) con el siguiente esquema: 1 minuto a 95ºC, 40 ciclos de 1 minuto a 95ºC, 1 minuto a 50ºC, 1 minuto a 72ºC, finalmente 1 minuto a 72ºC. Además, los productos amplificados se corrieron en un gel de agarosa al 2% y teñido con bromuro de etidio. Los productos se visualizaron en un analizador de imágenes.
Medios que se utilizaron
Los medios utilizados para crecer los cultivos celulares recibieron el mismo esquema de trabajo. Como control positivo se utilizó la cepa ATCC Mycoplasma fermentans PG-18. Asimismo, la comparación de los resultados obtenidos entre el método microbiológico y PCR se realizó con la prueba de T de Student.
Resultados
Un total de veinte muestras de diferentes cultivos celulares y veinte muestras de medios utilizados frecuentemente para crecer los cultivos celulares fueron analizadas para detectar presencia de micoplasmas. Por el método microbiológico se detecto en 9/20 muestras (45%) presencia de micoplasmas en los cultivos celulares, en los medios utilizados para crecer los cultivos celulares solamente en 2/20 muestras (10%) se detectó presencia de micoplasmas.
El cultivo y detección de micoplasmas fue por medio de medios específicos para estos microorganismos, en una primera etapa en fase líquida y en una segunda etapa en fase sólida, en esta última presentando la morfología característica (Figura 1).
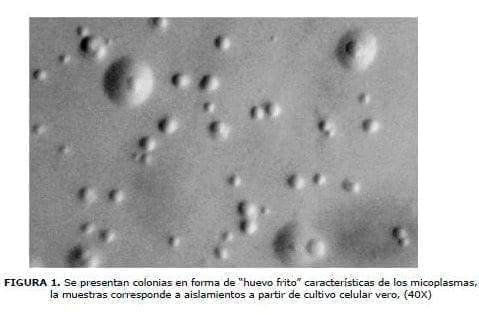
Al realizar la detección por medio de la prueba de PCR resultaron positivas 10/20 muestras (50%) de cultivos celulares y 2/20 muestras (2%) de los medios utilizados para crecer los cultivos celulares (Tabla 1).
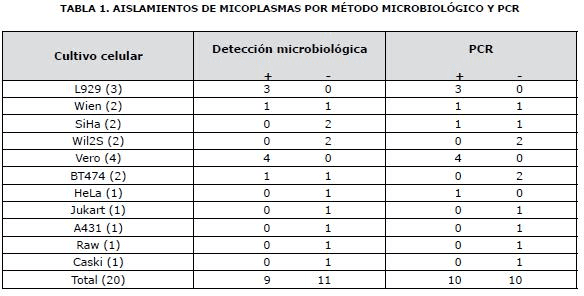
El análisis estadístico mostró que no hay diferencia significativa (P>0.05) en la detección entre el método microbiológico y PCR.
Discusión
El haber incluido los medios utilizados para crecer los cultivos celulares permite descartar la contaminación por estos y no por un mal manejo por parte del operador, sin embargo se observó que un 2% de las muestras de los medios utilizados estaban contaminados por micoplasmas.
Con respecto a los cultivos celulares, estos presentaron 45% y 50% del total contaminados por medio del método microbiológico y PCR respectivamente. Reportes de los últimos cuatro años muestran entre un 61.2% y 88.7% de contaminación por micoplasma en cultivos celulares (8,9).
El cuadro 1 presenta que el cultivo celular SiHa y HeLa son negativos a presencia de mi coplasmas por el método microbiológico, sin embargo por la técnica de PCR se validó una muestra en ambos cultivos celulares.
Y por el contrario en los cultivos celulares BT474 que mostró 1 de 2 muestras positivas a micoplasmas por el método microbiológico, con la técnica de PCR las dos muestras resultaron negativas, lo anterior puede estar en relación a la mínima cantidad de muestra que no fue suficiente para evidenciar la presencia de micoplasmas por esta técnica , ya que se ha reportado discrepancia en los resultados, interpretándose como falsos negativos o falsos positivos en base a la comparación de distintos métodos de detección.
De tal forma se ha reportado que la detección de micoplasmas por el método microbiológico, técnica de hibridación de ADN y por PCR son susceptibles a producir resultados falsos negativos o falsos positivo, de tal forma sugerimos que se deben implementar al menos dos técnicas para validar los resultados durante la búsqueda de contaminación por micoplasmas en cultivos celulares (10-14).
En conclusión, los datos presentados permiten plantear que la detección de micoplasmas en cultivos celulares debe reforzarse con otra técnica para validar los resultados.
Agradecimientos y financiación
A la Vicerrectoria de Investigación y Estudios de Posgrados de la Benemérita Universidad Autónoma de Puebla por el financiamiento otorgado al proyecto RITJ-NAT10-I y la beca otorgada a Linda Valeria Castillo-Viveros.
Conflictos de intereses
Ninguno que declarar.
Referencias bibliográficas
1. Rivera-Tapia JA, Cedillo-Ramírez ML, Gil-Juárez C. “Some biological features of mollicutes” Rer Latinoam Microbiol 2002; 44: 53-57.
2. Mayo CDM, Barrios BV, Ruiz AR, Cedillo RL, Rivera TJA. “Aislamiento de mollicutes en faringe y tracto urogenital.” 2009; Enf Inf Microbiol; 29: 6-10.
3. Baseman JB, Tully JG. “Mycoplasmas: sophisticated reemerging, and burdened by their notoriety” Emerg Infect Dis 1997; 3: 21-32.
4. Nicolson GL, Gan R, Haier J. “Multiple co-infections (Mycoplasma, Chlamydia, human herpes virus-6) in blood of chronic fatigue syndrome patients: association with signs and symptoms”. APMIS 2003; 111: 557-566.
5. Miyazaki K, Takaku H, Umeda M, Fujita T, Huang WD, Kimura T. “Potent growth inhibition of human tumor cells in culture by arginine deiminase purified from a culture medium of a mycoplasma-infected cell line” Cancer Res 1990; 50: 4522-4257.
6. Chambaud I, Wroblewsaki H, Blanchard A. “Interactions between mycoplasma lipoproteins and the host immune system”. Trends Microbiol 1999; 7: 493-499.
7. Garner CM, Hubbold LM, Chakraborti PR, “Mycoplasma detection in cell cultures: a comparison of four methods”. Br J Biomed Sci 2000; 57: 295-301.
8. Timenetsky J, Santos LM, Buzinhani M, Mettifogo E. “Detection of multiple mycoplasma infection in cell cultures by PCR”. Braz J Med Biol Res 2006; 39: 907-914.
9. Rivera A, Rivera E, Giono S, Gil C, Cedillo L. “Cell cultures contaminations by mycoplasmas”. Afr J Microbiol Res 2009; 3: 637-640.
10. Van Kuppeveld FJM, Johansson KE, Galama JMD, Kissing J, Bolske G, Van Der Logt, Melchers WJG. “Detection of mycoplasma contamination in cell cultures by a mycoplasma group-specific PCR”. Appl Environ Microbiol 1994; 60: 149-152.
11. Kong F, James G, Gordon S, Zelynski A, Gilbert GL. “Species-specific PCR for identification of common contaminant mollicutes in cell culture” Appl Environ Microbiol 2001; 67: 3195-3200.
12. Sung H, Kang SH, Bae YJ, Hong JT, Chung YB, Lee CK, Song S. “PCR-based detection of mycoplasma species”. J Microbiol 2006; 44: 42-49.
13. Schmitt M, Pawlita M. “High-throughput detection and multiplex identification of cell contaminations”. Nucleic Acids Res 2004; 37e: 119-126.
14. Stömer M, Vollmer T, Henrich B, Kleesiek K, Dreier J. “Broad-range real-time PCR assay for the rapid identification of cell-line contaminations and clinically important mollicute species”. Int J Med Microbiol 2009; 299: 291-300.