La cepa 125H fue empleada para seleccionar cual de los iniciadores permitia obtener el mejor RAPD tipo. Este ensayo demostró que empleando el iniciador OPA-07 se obtiene la mejor distribución de las bandas polimórficas del ADN, resultado que se esperaba con los iniciadores con mayor por ciento de guanina y citosina (figura No 1).
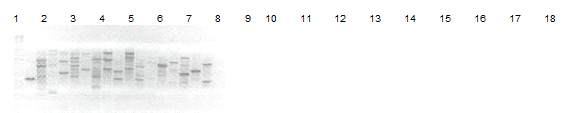
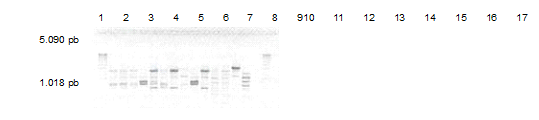
Una vez seleccionado el iniciador OPA-07 se utilizó para investigar la relación genética entre los aislamientos de O157:H7. Este análisis reportó la presencia de 1 banda para el caso del aislamiento 18N y 15 bandas para los aislamientos 20H y 132 H, con tallas moleculares que oscilaban entre los 1,018 y los 5,09 pb (figura No 2).
La tipificación resultante del análisis por RAPD demuestra la variación intraserotípica como resultado de la presencia de al menos una banda polimórfica de ADN. Estas diferencias permitieron identificar 11 RAPD tipos, que fueron designados con la letra mayúsculas C, seguida de un número arbitrario que permitió diferenciar los RAPD tipos (Tabla No 2). Nueve de estos RAPD (81.8%) fueron exclusivos de algunos aislamientos, sin embargo, los dos patrones más comunes de RAPD (18.18%) abarcaron 5 de los 14 aislamientos. La distancia genética entre los patrones de RAPD, calculada en por ciento estuvo entre 0.04 y 0.79 (tabla No 3), (figura No 3).
Tabla No 2. Número de bandas polimórficas y RAPD
tipos encontrados entre las cepas de EHEC O157:H7

Tabla No 3. Distancia genética expresada en por
ciento entre los RAPD tipos de E.coli 0157:H7.

S (Coeficiente de Jacckard)

Figura No. 3 Dendograma derivado del análisis de los RAPD tipos de los aislamientos de
E. coli 0157:H7. Los valores de la escala indican la distancia genética en por ciento
Discusión
Este estudio demuestra que el “primer” (cebador o iniciador) OPA-07 seleccionado puede ser utilizado para amplificar los fragmentos polimórficos del ADN de cepas diferentes de ECEH O157:H7. Al hacer una comparación entre los resultados obtenidos con los cebadores con un contenido de C+G superior al 50% (figura No 1), es factible concluir que la secuencia del cebador juega un papel primordial en la caracterización. En 1995 Madico et al.10, reportaron la obtención de diferentes RAPD tipos de E.coli O157:H7 con alta reproducibilidad a través del uso de cinco cebadores con más de 50% de C+G, lo cual permitió generar bandas en un rango aproximado entre 0,3 y 3,1 Kb. En este estudio hemos obtenido resultados similares empleando el cebador OPA-07, el que permitió generar bandas en un rango entre 1,018 y 5,09 bp.
Entre los resultados más importantes de esta parte del estudio están: primero, haber encontrado una marcada heterogeneidad entre los aislamientos de O157:H7, heterogeneidad representada por el hallazgo de 11RAPD tipos distintos durante el estudio. Segundo, que la distancia genética representada en el dendograma indica que las cepas de O157:H7 están agrupadas en dos “cluster” (grupos) mayoritarios, el primero representado por los RAPD tipos C2, C3, C4, C5, C6, C7, C8, C9, C10 y C1, el segundo representado por el RAPD tipo C1 (figura No 3), (tabla No 2).
Comparando los resultados obtenidos con la técnica de RAPD y la electroforesis de campo pulsado (PFGE), técnica que se le compara en cuanto a sensibilidad11, encontramos que para la tipificación de O157:H7 por PFGE se emplea la endonucleasa de restricción Xba I, lo que permite generar más de 20 fragmentos polimórficos12,13,14,15. En nuestro caso el análisis de RAPD generaron hasta 15 bandas polimórficas demostrando una eficiencia y una sensibilidad similar con relación al PFGE. Otros autores han reportado la tipificación de diferentes serotipos de E.coli enterotoxigénica (ECET) empleando el RAPD y diferentes cebadores con más del 50% en contenido de C+G, logrando 4, 8 y hasta 12 fragmentos polimórficos dependiendo de la secuencia del cebador8; en estos estudios no se aclara si estos fragmentos son 100% polimórficos o si han estado presentes en más de una de las cepas caracterizadas en el estudio. En nuestro caso es factible afirmar que el número de bandas encontradas en cada RAPD depende únicamente de cada aislamiento, por el hecho de haber empleado un único primer; por otra parte se encontraron 10 bandas o fragmentos polimórficos amplificados, repetidos como máximo en dos aislamientos diferentes.
Durante un período de un año que demoró el estudio, 9 de los 14 pacientes pediátricos (64.3%) identificados como portadores de E.coli O157:H7 presentaron un único RAPD tipo. Sin embargo, en el 35.7% de los pacientes pediátricos (5 pacientes) portadores de E.coli O157:H7 se encontraron RAPD tipos repetidos al menos una vez. Es importante destacar la ausencia de una asociación epidemiológica entre los aislamientos. No obstante, la técnica de RAPD reveló variaciones intraserotípicas específicas entre las cepas de E.coli O157:H7; variaciones que no son detectables aún con la combinación de diferentes técnicas de caracterización fenotípica.
Los resultados obtenidos en este trabajo son de gran importancia epidemiológica, e indican que un número limitado de ECEH O157:H7 genéticamente cercanas han estado circulando en los hospitales de Bogotá durante un período de al menos un año.
Adicionalmente un estudio realizado empleando la técnica de electroforesis de campo pulsado (PFGE) demuestran que en Bogotá están circulando varios clones no muy distantes genéticamente; sin embargo, existe una aparente infección cruzada entre bovinos, productos cárnicos y humanos contaminados con E.coli O157:H7 (datos no mostrados).
Es cierto que el número de cepas de ECEH analizadas en este estudio no constituyen una colección representativa de la zona donde se llevaron a cabo los aislamientos y que el grado de diversidad genética está subestimado, pero resulta indudable que en una colección reducida de O157:H7 y restringida geográficamente como la nuestra, se ha encontrado una diversidad genética considerable (RAPD tipos), gracias al empleo de la técnica de RAPD.
Bibliografía
- Máttar S, Mora A ,Bernal N. Prevalencia de E. coli O157:H7 en una Población Pediátrica de Bogotá D.C. con Gastroenteritis Aguda. Enferm. Infecc Microbiol Clin 1997; 15:364-8.
- Máttar S, Vásquez E. Escherichia coli O157:H7 Infection in Colombia. Emerg Infect Dis 1998; 4:126-7.
- Máttar S, Poutou RA, León Y, Murcia S. Seguimiento de las Infecciones por Escherichia coli O157:H7 Aisladas de Distintas Fuentes A Través del Análisis Plasmídico. Medicas UIS 1998; 12:285-8.
- Máttar S, Benavides M, Alarcón S, Garzón L, et al. Investigación Epidemiológica sobre la Prevalencia de Escherichia coli O157:H7 en Humanos, Bovinos y Productos Cárnicos en Colombia. Enferm Infec Microbiol 1998; 18:45-50
- Willians JGK, Kubelik AR, et al, DNA Polymorphic Amplified by Arbitrary Primers are Useful as Genetic Markers. Nucl Acid Res 1990; 18:6531-5.
- Maslow JN, Mulligan ME, Arbeit RD. Molecular Epidemiology: Applicacion of Conteporary Techniques to the Typing of Microorganisms. Clin Infec Dis 1993; 17:153-64.
- Banatvala N, Magnano AR, Cartter ML, et al. Meat Grinders and Molecular Epidemiology: Two Supermarket Outbreaks of Escherichia coli O157:H7 Infection. J Infect Dis 1996; 173: 480-483.
- Pacheco AB, Guth B, Soares KCC. et al. Random Amplification of Polymorphic DNA Reveals Serotype-Specific Clonal Cluster Among Enterotoxigenic Escherichia coli Strains Isolated from Humans. J Clin Microbiol 1997; 35:1521-5
- Sambrook J, Manniatis T, Fritsch EF. Molecular Cloning: A Laboratory Manual. Manniatis T and Sambrook Editors. Cold Spring Harbor Laboratory Press Edit Second Edition. New York: 1989
- Madico G, Akopyants N, Berg DE. Arbitrarily Primer PCR DNA Fingerprinting of Escherichia coli O157:H7 Strains by Using Templates from Boiled Cultures. J Clin Microbiol 1995; 33: 1534-6
- Wang G, Whittam C, Berg M, Berg DE. 1993. RAPD (arbitrary primer) PCR Is More Sensitive than Multilocus Enzyme Electrophoresis for Distinguishing Related Bacterial Strains. Nul Acid Res 1993; 21:5930-3
- Johnson J, Weagant S, Jinneman K, Bryant J. Use of Pulsed – Field Gel Electrophoresis for Epidemiological Study of Escherichia coli 0157 :H7 During A Food Borne Outbreak. Appl Environ Microbiol 1995; 61: 2806-8.
- Krause U, Thomson-carter FM, Pennington TH. Molecular Epidemiology of Escherichia coli O157:H7 by Pulsed-Field Gel Electrophoresis and Comparison with That by Bacteriophage Typing. J Clin Microbiol 1996; 34:959-61
- Meng J, Zhao S, Zhao T. et al. Molecular Characterization of Escherichia coli O157:H7 Isolates by Pulsed-Field Electrophoresis and Plasmid DNA Analysis. J Med Microbiol 1995; 42:258-63
- Izumiya H, Terajima J, Wada A. et al. Molecular Typing of Enterohemorrhagic Escherichia coli O157:H7 Isolates in Japan by Using Pulsed-Field Gel Electrophoresis. J Clin Microbiol 1997; 35:1675-80
