FcgRI
Las estructuras y características básicas de los FcgR se presentan en la Figura 1 y la Tabla 1, respectivamente.

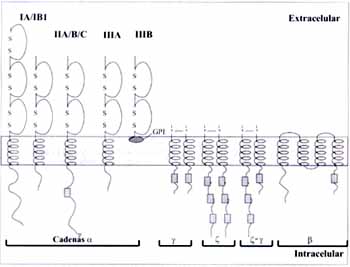
Figura 1.Estructura básica de los FcgR y de sus cedenas accesorias
Se han identificado tres genes que codifican para el FcgRI, son FcgRIA, B y C, los cuales están ubicados en el brazo largo del cromosoma uno, banda q21.1.
Estos genes consisten en seis exones, de los cuales los dos primeros codifican para el péptido señal, los exones tres, cuatro y cinco codifican los dominios tipo inmunoglobulina de la porción extracelular y finalmente el exón seis codifica para las porciones transmembrana e intracitoplasmática (8, 9).
El FcgRIA (CD64) es una glicoproteína de 72 kD que se une a la IgG monomérica con alta afinidad (Kd 10–10 M) (10). Tiene una región extracelular de 292 aminoácidos, constituida por tres dominios tipo Ig; además posee una región transmembrana de 21 aminoácidos y una intracitoplasmástica de 63 aminoácidos (Tabla 1).
El tercer dominio o amino terminal de la porción extracelular es el que confiere la alta afinidad a este receptor, diferencia fundamental con los receptores de baja afinidad (FcgRII y FcgRIII) que solo poseen dos dominios tipo Ig (11) (Figura 1).
Tabla 1. Características generales de los FcgR humanos
FcgRI (CD64) |
FcgRII (CD32) | FcgRIII (CD16) | |
| Ubicación genómica Genes TranscriptosAlelos Peso Molecular Afinidad |
1q21 A,B,C IA, IB1* IB2, IC 72 kD Alta (10 8 – 10 9 M-1 ) |
1q23-24 A,B,C IIA1, IIA2, IIB1, IIB2, IIB3, IICIIA: 131 LR/HR, 27 Q/W 40 kD Baja (10 7M-1) |
1q23-24 A,B IIIA, IIIB IIIA: 158 F/V, 48 L/R/H IIIB: NA1, NA2, SH 50 – 80 kD IIIA: Media (3X10 7M-1) IIIB: Baja ( <10 7M-1) |
| Expresión constitutiva
Publicidad Expresión inducida |
Monocitos, macrófagos células dendríticas, neutrófilos, eosinófilos células progenitoras mieloides Neutrófilos (G-CSF, IFN-g) Monocitos (IFN-g, IL-10) Mastocitos (IFN-g) |
IIA: Neutrófilos, monocitos, macrófagos, células dendríticas, plaquetas, algunas células T, células dendríticas, plaquetas, algunas células endoteliales IIB: Células B IIC: Desconocida Eosinófilos (IFN-gIL-3) |
IIIA: macrófagos, monocitos, células NK, algunas células T, mastositos IIIB: neutrófilos,eosinófilos Neotrófilos(IFN-gGM-CSF,G-CSF) Eosinófilos (IFN-g) Monocittos (TGFg b) |
| Asociación concadenas accesorias de señalización | Cadena g | Cadena g | Cadena b,g,V |
| Especificidad por IgG humana | 3>>1>4>>>2 | IIA R131: 3>1>>>2,4 IIA H131: 3>1,2>>>4IIB: 3>1>4>>2 |
1,3>>>2,4* |
*Receptores solubles
*El FcgRIIIA V158 tiene una afinidad más alta por La IgG1,3 y 4 que el alelo FcgRIIIA F158. El alelo FcgRIIIA NA1 tiene
Figura 1. Estructura básica de los FcgR y de sus cadenas accesorias.
El gen FcgRIA codifica para un transcripto único que se expresa como la proteína antes descrita. A diferencia de éste, los genes B y C contienen codones de parada en el tercer exón, lo cual permite que se originen variantes moleculares a partir de un mismo gen.
Por ejemplo, el gen B codifica dos transcriptos: FcgRIB1 y FcgRIB2. Este último aparece como una proteína trans-membrana con dos dominios tipo Ig, debido a que ocurre un corte y empalme alternativo durante el procesamiento del ARNm. Debido a estos cambios la proteína carece del primer dominio tipo inmunoglobulina que es el que confiere la alta afinidad por la IgG monomérica (10).
De otro lado, el transcripto B1, así como el trascripto del gen C, FcgRIC, no realizan procesamiento alternativo, lo cual da origen a transcriptos cortos que se traducen en FORMAS solubles de las proteínas FcR (Figura 2).
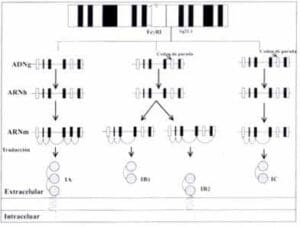
Figura 2. Localización cromosómica, corto y empalme alternativo y expresión del FcgRI.
La estructura básica de los FcgR consiste de una cadena a con una porción extracelular compuesta por 2 o 3 dominios tipo Ig, una región transmembrana y otra intracitoplasmática. El FcgRIIIB no tiene región transmembrana y tampoco cola intracitoplasmática y se une a la membrana por medio de una molécula glicosilfosfatidilinositol (GPI). Las cadenas g y z pueden estar presentes en la membrana como homodímeros o heterodímeros y participan de la señalización intracelular mediada por los FcgR. La cadena b funciona como un amplificador de esta señalización (1).
Los cuadros grises representan los dominios de activación de inmunorreceptores basados en tirosina (ITAM).
Debe recordarse que el FcgRIIB posee en su cola intracitoplasmática un dominio de inhibición de inmunorreceptores basados en tirosina (ITIM), el cual modula negativamente las respuestas mediadas por los ITAM de los otros receptores.
Se muestra la ORGANIZACIÓN cromosómica exón-intrón de los genes FcgRIA, B y C. Los exones se representan con rectángulos y los intrones con líneas. Los rectángulos negros representan las secuencias que después de la traducción hacen parte de la proteína madura, los rectángulos no sombreados representan regiones que no se expresan en la proteína madura.
El FcgRIA y el FcgRIB se expresan en la membrana celular. Los receptores solubles se generan por distintos mecanismos que incluyen: codones de parada en el dominio extracelular y corte y empalme alternativo del ARNm.
(Lea También: FcgRII: Receptores FC de la Inmunoglobulina)
ADNg: ácido desoxiribonucleico génomico; ARNh: ácido ribonucleico heterogéneo; ARNm: ácido ribonucleico mensajero
El FcgRIA se expresa en la membrana celular asociado a la cadena g. Esta asociación es necesaria para que este receptor tenga una expresión y función adecuada (12-14).
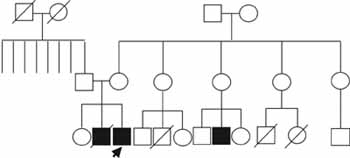
Aunque el FcgRIA tiene un papel importante en el sistema inmune, su presencia parece no ser totalmente indispensable. Recientemente se reportó una familia en la que varios de sus miembros presentaban ausencia del FcgRIA y pese a esto eran aparentemente saludables (15).
La molécula FcgRIA tenía una expresión 15 a 20 veces menor en los monocitos de estos individuos en comparación con el grupo control.
La diferencia encontrada en estos pacientes se debió a un cambio de un solo nucleótido (C —->T) en el exón 1, el cual daba origen a un codón prematuro de terminación en la posición aminoacídica 92, lo que a su vez predecía la traducción de un péptido corto y no funcional; además se demostró que este cambio afectaba la estabilidad del ARNm (16).