Articulo de investigación: Estudio de Variantes Moleculares de los Genes PTPN22, TNF Y VDR en Madres de Niños con Nefritis Lúpica
Asociación como Factores de Riesgo
Gloria Garavito de Egea1, Eduardo A. Egea Bermejo2, Clara Malagón3, Luis Fang Mercado4, Carlos Olmos5, Luz González6, Pilar Guarnizo7, Fernando R. De la Cruz López8, Gustavo Aroca9, Guillermo López Lluch10, Antonio Iglesias11
Resumen
La Nefritis Lúpica (NL) es más frecuente en mujeres. Se reconoce que madres con Lupus Eritematoso Sistémico (LES) confieren mayor riesgo al desarrollo de esta entidad en sus hijos. Esta transmisión se debe a la impronta genómica y/o al genotipo materno sobre el desarrollo prenatal.
En este estudio se identificaron variantes de los sistemas PTPN22, VDR y TNF, asociadas a NL pediátrica (NLp). Se investigó la asociación de marcadores en 64 familias: 46 tríos (Caso/Padre- Madre) y 18 dúos (Caso/Madre).
Se genotipificaron los SNPs rs2476601 [A/G] de PTPN22; rs361525 [A/G] y rs1800629 [A/G] de TNF; TaqI [rs731236 A/G], ApaI [rs7975232 A/C], BsmI [rs1544410 C/T] y FokI [rs2228570 A/G] de VDR mediante RT-PCR. Se estimó el efecto de la sobretransmisión del alelo de riesgo de padres a hijos.
Se estimó el efecto genético de los SNPs sobre los niños (R1 y R2) y también se estudiaron la infl uencia genética materna (S1 y S2) y la impronta materna (Im). Se observó que el alelo A de rs2476601 en PTPN22 es sobretransmitido (p=0,028) en los niños con nefritis lúpica y se demostró que los niños portadores de una copia del alelo A de rs2476601 presentan un riesgo (R1) de 0,20, mientras que dos copias del alelo (R2) lo incrementan a 1,71.
Además, si la madre es portadora de dos copias del alelo A (S2), el riesgo aumenta a 2,5. La impronta genética fue de 0,97 (p=0,002). Nuestro estudio describe la infl uencia materna de las variantes de PTPN22, TNF y VDR sobre niños con NLp en familias colombianas.
Palabras clave: Lupus eritematoso sistémico, Nefritis Lúpica, Epigenética, Impronta genómica, Modelos Genéticos. (Vea también: Artículo de Investigación: Niños Obesos del Caribe Colombiano, Un Estudio de Casos y Controles)
Maternal Genetic Variants in Ptpn22, Tnf And Vdr and the Risk of Pediatric Lupus Nephritis
Abstract
Lupus nephritis (LN) is more common in women. It is recognized that mothers with SLE confer increased risk to develop this entity to their children. This transmission is due the genomic imprint and/or maternal genotype on prenatal development. In this study, variants of PTPN22, TNF and VDR genes were identifi ed and associated with pediatric LN (PLN).
The association of markers was investigated in 64 families: 46 trios (case/Father-Mother) and 18 duos (case/ Mother). The SNPs rs2476601 [A/G] of PTPN22; the rs361525 [A/G], rs1800629 [A/G] from TNF and TaqI [rs731236 A/G], ApaI [rs7975232 A/C], BsmI [rs1544410 C/T] and FokI [rs2228570 A/G] of VDR gene were genotyped by RT-PCR.
The effect of over-risk allele transmission from parents to children was estimated. The genetic effect of the SNPs on children (R1 and R2) was estimated and maternal genetic infl uence (S1 and S2) and maternal imprinting (Im).
It was observed that the A allele of rs2476601 in PTPN22 is over-transmitted (p = 0,028) to PLN children, and that children carrying one copy of the allele of rs2476601 have a (R1) risk of 0,20; while two copies of allele (R2) increase it to 1,71.
Also, if the mother carries two co- pies of allele A (S2), the risk become 2,5. DNA fi ngerprinting was 0,97 (p = 0,002). Our study describes maternal infl uence of the variants of PTPN22, TNF and VDR genes on children with PLN in colombian families.
Key words: Systemic Lupus Erythematosus, Lupus Nephritis, Epigenetic Genomic Imprinting, Genetic Models.
Introducción
El Lupus Eritematoso Sistémico (LES) es una enfermedad autoinmune compleja, caracterizada por la producción de autoanticuerpos contra moléculas nucleares, citoplasmáticas y de la superfi cie celular de quien la padece (1-3).
Sus manifestaciones comprenden una amplia gama de signos y síntomas clínicos como erupciones cutáneas, fotosensibilidad, úlceras orales, artritis, pleuritis, pericarditis, nefropatías y convulsiones (4). Se desconocen las causas de LES; sin embargo, se ha demostrado su vínculo con factores genéticos y ambientales (5-7).
Por su parte la nefritis lúpica (NL) es un en- dotipo de LES que afecta a tres de cada 100.000 personas (8). Previamente se ha demostrado la asociación de variantes genéticas presentes en los genes de PTPN22, VDR, TNF y HLA en adultos con endotipo de Nefritis Lúpica tipo IV (NL IV); así como la aparición a temprana edad de Nefritis Lúpica Pediátrica (NLP).
Como se ha descrito en la literatura, esta enfermedad autoinmune ha demostrado un comportamiento sexodependiente, donde el género femenino presenta 2,6 (95%IC: 1,38 – 4,91) y 2,73 (95%IC: 1,22 – 6,11) veces más probabilidad de padecer NLP y NL IV, respectivamente en poblaciones de la región caribe colombiana.
La evidencia confirma que LES afecta predominantemente a las mujeres más que a los hombres, en una proporción de 6-10, especialmente a temprana edad (9,10). Además, se ha descrito que madres con diagnóstico de LES confieren mayor riesgo al desarrollo de la enfermedad en sus hijos que los padres (11,12).
Aunque algunos reportes en la literatura no demuestran coherencia en los resultados, en la mayoría de los casos por las diferencias entre las poblaciones estudiadas es probable que el abordaje del estudio genético sea insuficiente para responder interrogantes sobre el desarrollo de la misma (13,14).
Los estudios de asociación de casos y controles son una alternativa ampliamente usada para la identificación de factores genéticos de riesgo sobre una enfermedad en particular (15, 16). Sin embargo, este enfoque metodológico ignora mecanismos adicionales que influyen sobre la enfermedad.
Las bases moleculares de esta transmisión preferencialmente materna de riesgo de LES son actualmente desconocidas, pero se puede responder mediante dos mecanismos biológicos: la impronta genómica y el efecto directo de genotipo materno (11,17).
En la impronta genómica el alelo heredado (ya sea del padre o de la madre) se expresa, mientras que el alelo alterno es silenciado. Así el efecto de un alelo dependerá de su origen (padre o madre) resultando con diferencias fenotípicas entre los heterocigotos con translocaciones recíprocas (efecto de origen paterno o materno) (18).
Recientemente se ha demostrado que los efectos de origen paterno en enfermedades complejas se deben a variaciones genéticas en los genes impresos (19). Por su parte los efectos de genotipo materno ocurren cuando el genotipo materno influye directamente sobre el fenotipo de la descendencia.
Este efecto es completamente independiente del genotipo propio de la descendencia y ocurre a través del ambiente proporcionado por la madre durante el desarrollo prenatal. El efecto del genotipo materno puede conllevar diferencias fenotípicas entre heterocigotos recíprocos, considerándose de este modo como efectos de origen parental específico (17, 20).
Nuestra pregunta científica se orientó a determinar si las variantes genéticas en los loci PTPN22, VDR y TNF pueden dar respuesta al comportamiento génerodependiente que se observa en LES a partir de la impronta genómica y efectos de origen materno.
Para esto se realizó un estudio de tríos (Caso/Padre- Madre) y dúos (Caso/Madre), en donde el caso índice correspondió a un niño(a) con diagnóstico clínico y anatomopatológico de Nefritis Lúpica Pediátrica.
Métodos
Población de estudio
Se realizó un estudio analítico para determinar la asociación de polimorfismo tipo SNPs de tres sistemas genéticos: PTPN22, TNF y VDR en una cohorte de 64 familias colombianas: 46 de ellas fueron tríos (Caso/Padre-Madre) y 18 dúos (Caso/ Madre) para un total de 174 individuos en los cuales se identificaron marcadores de impronta genómica materna.
En este estudio solo se incluyeron familias cuyos padres (papá/mamá) no refirieron antecedentes personales de enfermedades autoinmunes. A todos los participantes se les tomaron muestras de sangre periférica para la extracción de ADN genómico, previo asentimiento y consentimiento informado por parte del menor de edad y el padre o tutor legal respectivamente. El presente estudio contó con aprobación del Comité de Ética de la Universidad del Norte.
Genotipificación de SNPs
El ADN genómico se extrajo a partir de leucocitos de sangre periférica mediante la técnica de Salting-out (21). Se genotipificaron los SNPs rs2476601 [A/G] en el gen de la Proteína Tirosina Fosfatasa no Receptora Tipo 22 (PTPN22); rs1800629 [A/G] y rs361525 [A/G] del gen del Factor de Necrosis Tumoral alfa (TNF-α); y los polimorfismos TaqI [rs731236 A/G], ApaI [rs7975232 A/C], BsmI [rs1544410 C/T] y FokI [rs2228570 A/G] presentes en el gen del receptor de vitamina D (VDR).
Estos SNPs se estimaron mediante PCR en tiempo real (RT-PCR) utilizando estuches disponibles comercialmente TaqMan® SNP Genotyping [Assay ID: C__16021387_20, C___7514879_10, C___2215707_10, C___2404008_10; C__28977635_10; C__12060045_20 y C___8716062_10; respectivamente] de la casa Applied Biosystems, Foster City, CA, USA.
Los ensayos de genotipificación se realizaron en un equipo de RT-PCR ABI Prism 7300, de Applied Biosystems con un volumen total por reacción de 5μL (2.4μL de DNA a una concentración aproximada de 10ng/μL, 2.5μL de Master Mix-2x y 0.125μL de sondas Taqman Genotyping-40x específicas de cada SNP). Todas las reacciones de PCR se realizaron por duplicado.
El programa de ciclado consistió de un paso inicial de 10 minutos a 95°C, seguido por 40 ciclos de 15 segundos a 92°C y 1 minuto a 60°C por ciclo. La asignación genotípica se realizó automáticamente mediante la aplicación de Allelic Discrimination del sistema Applied Biosystems, la cual tuvo en cuenta una calidad de amplificación ≥90% por muestra.
Análisis estadístico
El análisis estadístico consistió en determinar las frecuencias alélicas y genotípicas mediante el software estadístico SPSS v20 (IBM® SPSS® Statistics 20; IBM Corp., USA). A estas frecuencias se les estimó la desviación del equilibrio de Hardy-Weinberg y se compararon entre los padres y descendientes usando el Test de diferenciación de muestras de Arlequin v3.5.
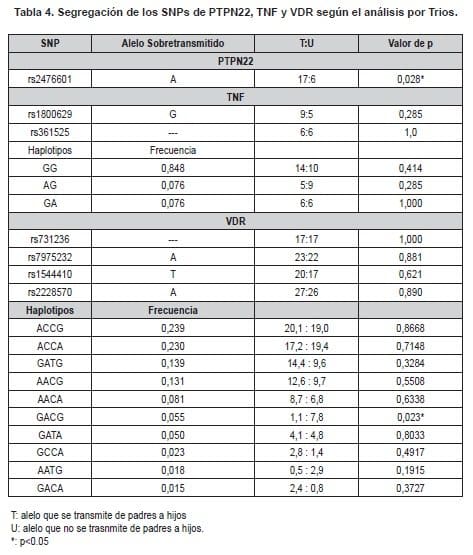
La segregación de los SNPs de PTPN22, VDR y TNF entre los Tríos, se realizó mediante el cálculo de los estadísticos de desequilibrio de ligamiento (LD) como son el D´ y el r2, así como la estimación de los alelos de riesgo para cada SNP que eventualmente se sobretransmite de padres a hijos; para estos análisis se empleó el software Haploview 4.1.
En el análisis de familias también se estimó el efecto genético de los SNPs de PTPN22, VDR y TNF sobre los niños (R1 y R2), el efecto genético materno (S1 y S2) y la impronta materna (Im) teniendo en cuenta todas las familias mediante las herramientas de análisis genético PREMIM-EMIM (22, 23).
Estos fenómenos se estimaron a partir de modelos de probabilidad multinomial, asumiendo que los padres del estudio se encuentran en equilibrio genético de Hardy-Weinberg y existe apareamiento aleatorio en esta.
La probabilidad de riesgo de la enfermedad en los niños en caso de presentar una copia del alelo de riesgo se representa como “R1” y si presenta dos copias del alelo de riesgo se representa como “R2”, el efecto materno es representado como “S1” (si la madre porta una copia del alelo de riesgo) y “S2” (si la madre porta dos copias del alelo de riesgo); la impronta genética es descrita como “Im”; todos estos parámetros fueron estimados en un modelo genético. La significancia estadística fue interpretada como p<0,05.
1 MD-Ph.D, Universidad del Norte, División Ciencias De la Salud, Grupo de Investigación en inmunología y Biología Molecular, Barranquilla-Colombia. 2 MD-Ph.D, Universidad del Norte, División Ciencias De la Salud, Grupo de Investigación en inmunología y Biología Molecular, Barranquilla-Colombia. 3 MD-Especialista, Universidad del Bosque, Ciencias de la Salud, Bogotá-Colombia. 4 MSc, Corporación Universitaria Rafael Núñez. Universidad de Cartagena. Cartagena-Colombia. 5 MD-Especialista, Universidad del Rosario, Ciencias de la Salud, Bogotá –Colombia. 6 MD-Especialista, Universidad del Bosque, Ciencias de la Salud, Bogotá-Colombia.
7 MD-Especialista, Universidad del Rosario, Ciencias de la Salud, Bogotá –Colombia. 8 B.Sc(C), Universidad del Norte, División Ciencias de la Salud, Grupo de Investigación en inmunología y Biología Molecular, Barranquilla-Colombia. 9 MD-Especialista, Universidad Simón Bolívar, Ciencias de la Salud, Grupo de Investigación Nefrología, Barranquilla-Colombia. 10 B.Sc-Ph.D, Universidad Pablo de Olavide. Departamento de Fisiología, Anatomía y Biología Celular, Sevilla-España. 11 MD-Especialista, Universidad Nacional, Ciencias de la Salud, Grupo Unidad de Reumatología, Bogotá –Colombia.
Resultados
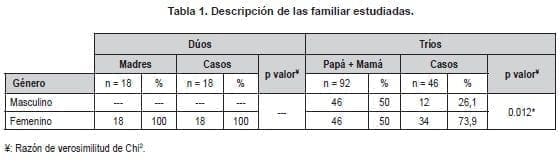
Se realizó un estudio de asociación genética en 64 familias de la región Caribe colombiana, 46 de ellas fueron tríos constituidos por familias nucleares (papá, mamá e hijo) y 18 dúos (madre e hijo). Los niños con un promedio de edad de 16,19 ± 3,48 años, contaron con diagnóstico de LES endotipo de Nefritis Lúpica, a diferencia de los padres, los cuales no reportaron antecedentes personales de alguna enfermedad autoinmune.
La distribución de la enfermedad demostró un comportamiento género dependiente, siendo más frecuente en niñas tanto en los dúos como tríos (100% [n=18] y 73,9% [n=34], respectivamente) (Tabla 1).
En el presente estudio se evaluaron variantes genéticas en los loci PTPN22, TNF, y VDR. En cuanto a la distribución genotípica y alélica de estas variantes en los dúos, se pudo observar que el SNP rs2476601 de PTPN22 no presenta variación, siendo el genotipo GG y alelo G los únicos presentes en el grupo de estudio.
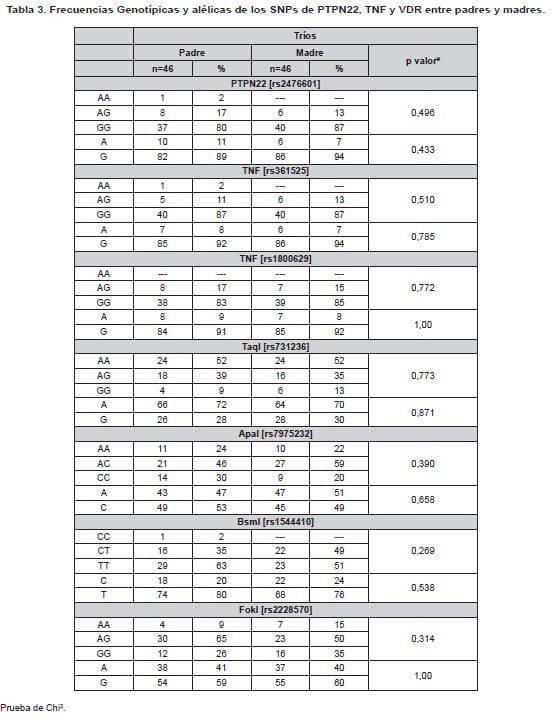
Sin embargo, en los tríos se observó el alelo A con una frecuencia del 8.69% [n=16] entre los padres y 19.56% [n=18] en la descendencia (p=0.017). Este polimorfismo en el grupo de niños no se encuentra en equilibrio genético de Hardy-Weinberg Los demás SNPs genotipificados no mostraron diferencias significativas en sus distribuciones, como se puede observar en la Tabla 2.
Por otra parte, no se observó diferencia significativa en cuanto a la distribución de los SNPs entre padres y madres (Tabla 3).
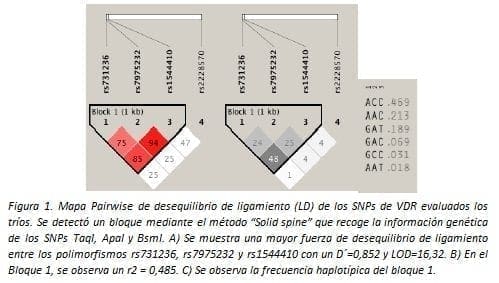
A partir de los tríos se estimó el desequilibrio de ligamiento de los SNPs de TNF y VDR entre los niños y los padres. Este análisis permitió identificar un bloque de 1kb de distancia genómica entre los SNPs rs731236, rs7975232 y rs1544410 mediante el método “Solid spine”, el cual abarca la región comprendida entre las posiciones 46.525.024 y 46.526.102 del cromosoma 12 correspondiente al gen de VDR.
Estos tres polimorfismos demostraron un desequilibrio significativo con un D´:0.807 y un r2=0.538 (Figura 1). Mientras que para los SNPs de TNF, también se observó un bloque de 3kb que incluye a los dos polimorfismos que se segregan conjuntamente (Figura 2).

Efecto directo de genotipo materno
La prueba para efectos de origen paterno se realizó con el software PREMIM-EMIM. Esta herramienta utiliza un enfoque de máxima probabilidad basado en modelos multinomiales para el modelado flexible de efectos genéticos a partir de datos de familias nucleares; sin embargo, en el presente estudio se aumentó el poder estadístico al incluir dúos familiares.
Para el análisis se realizó inicialmente un modelo genético básico, al cual sucesivamente se incluyeron parámetros de riesgo adicionales relacionados a efectos de origen paterno. El modelo complejo se construyó a partir del efecto del genotipo de los niños sobre su fenotipo de NL y el efecto del genotipo materno sobre el fenotipo de los niños si la madre es portadora del alelo de riesgo, además del efecto de la impronta genética.
En cuanto al efecto genético del SNP rs2676601 de PTPN22, no se observó un riesgo relativo de Nefritis Lúpica en los niños heterocigotos que cuentan con una copia del alelos de riesgo – alelo A – (R1: 0,20), mientras que en los niños homocigotos para el alelo A, quienes presentan dos copias del alelo de riesgo, el genotipo propio muestra un riesgo relativo de 1,71 (Parámetro R2).
En cuanto al efecto materno, el riesgo que tienen los niños de sufrir la enfermedad si la madre es portadora de una copia del alelo A es muy similar al parámetro descrito anteriormente (S1:0,5); sin embargo, si la madre es portadora de dos copias del alelo de riesgo (Parámetro S2) el riesgo aumenta a 2,5 veces. Por último la impronta genética no se comportó como un factor de riesgo relacionado con la enfermedad (Im: 0,97) (Tabla 5).
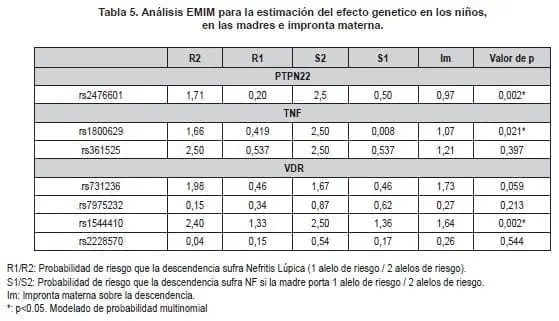
Es por esto que los niños homocigotos para el alelo de riesgo G presentan un riesgo relativo para el desarrollo de la enfermedad de R2:1,66 y este riesgo aumenta a 2,5 veces cuando la madre es portadora de dos copia del alelo (S2), a la vez que se observa la influencia de la impronta genética sobre la enfermedad en la descendencia con un riesgo relativo de 1,07 (Im) (Tabla 5).
De los tres loci evaluados, VDR mostró los mejores valores de riesgo para el desarrollo de Nefritis Lúpica en la descendencia. De los SNPs evaluados, el Bsml [rs1544410] mostró un riesgo relativo del alelo T 1,33 veces sobre el alelo C si los niños presentan una copia del alelo de riesgo (R1), y este riesgo aumenta a 2,4 cuando se presentan dos copias del alelo T (R2).
Incluso cuando la madre es homocigota para el alelo T la descendencia presenta 2,5 veces más riesgo de padecer la enfermedad (S2). Estos resultados son corroborados con el riesgo relativo arrojado por la impronta genética proveniente de la madre (lm: 1.64) (Tabla 5).
Discusión
Este es el primer estudio que reporta el efecto directo de genotipo materno con la susceptibilidad de Lupus Eritematoso Sistémico endotipo de Nefritis Lúpica sobre la descendencia independientemente del genotipo propio de los niños.
Este hallazgo fue encontrado en tres loci (PTPN22, TNF y VDR) que han mostrado su asociación genética con la enfermedad en diversos estudios previos (24,27). El efecto de origen paterno puede ser similar tanto para la impronta genética como el efecto de genotipo materno. Nuestros resultados sustentan el efecto directo de genotipo materno en los SNPs rs2476601 [A/G] de PTPN22, rs1800629 [A/G] de TNF y BsmI [rs1544410 C/T] de VDR.
No es claro cómo madres portadoras del o de los alelos de riesgo y sin diagnóstico de LES, pueden afectar el fenotipo de su descendencia. Nuestra hipótesis es que la respuesta inmune de la madre puede jugar un papel en el efecto materno; aún queda por dilucidar qué mecanismos inmunes participan en la NLP, en especial cuando factores ambientales condicionan la aparición de la enfermedad.
La importancia de la interacción madre-hijo durante la gestación conforma el fenotipo inmune del niño. Ejemplo es que infecciones durante el embarazo generan un mayor riesgo de trastornos en el neurodesarrollo, agravando la Encefalomielitis Autoinmune Experimental en la descendencia (28,29).
También hay reportes de niveles séricos de autoanticuerpos anti-Ro/SSA maternos, que se asocian con el desarrollo de bloqueo cardíaco o Fibroelastosis Endocárdica en neonatos con LES (30). A su vez datos experimentales han demostrado que la exposición uterina de anticuerpos o citoquinas maternas se convierte en factor de riesgo para el desarrollo de enfermedades del neurodesarrollo (31).
Por otra parte, se desconoce si la impronta inmunológica puede transmitirse de la madre al hijo; sin embargo, modificaciones epigenéticas pueden ser relevantes en la descendencia. Modificaciones como la metilación del ADN, modificación de las histonas y microRNA, son señales que regulan la expresión génica; estas son sensibles a los estímulos externos y podría ser el puente entre los factores genéticos y los factores ambientales (32).
Ejemplo es que el microRNA miR-1246 disminuye su expresión en células B activadas en pacientes con LES a través de la vía AKT-P53, aumentado a su vez la expresión de un Factor Temprano de células B (EBF1); por lo tanto, un cambio en la expresión de este microRNA puede interrumpir esta amplificación e inferir en la enfermedad en los niños (33).
No existen estudios que evalúen el efecto de origen paterno de las variantes genéticas de PTPN22, TNF y VDR. Sin embargo, se ha analizado el papel de HLA-DRB1 en relación con el efecto materno sobre descendencia con diagnóstico de LES (11).
Es notable la magnitud del efecto directo de genotipo materno en el SNP BsmI [rs1544410 C/T] de VDR (S1: 1,36 y S2: 2,5), pues este se ve complementado por la impronta genética proveniente de la madre con un riesgo de (lm: 1,64).
Las madres homocigotas para rs1800629 [A/G] de TNF y rs2476601 [A/G] de PTPN22, confieren a la descendencia un riesgo similar (S2: 2,5 y 2,5; respectivamente). Sin embargo, la impronta genética no es contundente debido a que muestran valores de riesgo relativamente bajos (Im: 1,07) y nulo (Im: 0,97). Esto demuestra la influencia del efecto directo del genotipo materno descrito en los tres SNPs sobre el LES.
A partir de lo anterior identificamos la influencia directa de los loci PTPN22, TNF y VDR de madres colombianas en la susceptibilidad de NLP en su descendencia, mediante análisis por familias (dúos y tríos). El efecto de genotipo materno reportado, es independiente de la herencia genética y puede deberse a su transmisión epigenética. Estos hallazgos deben ser validados con otros endotipos de LES.
Conflicto de Interés
Los autores del presente estudio declaran no tener conflicto de intereses en la realización del trabajo y del manuscrito.
Agradecimientos
Agradecemos a Colciencias (Código Colciencias: 121549362203) y a la Universidad del Norte por sus aportes en el financiamiento de esta investigación y nuestros pacientes por su colaboración.
Referencias
1. Mohan C, Putterman C. Genetics and pathogenesis of systemic lupus erythematosus and lupus nephritis. Nature reviews Nephrology. 2015.
2. Bonanni A, Vaglio A, Bruschi M, Sinico RA, Cavagna L, Moroni G, et al. Multi-antibody composition in lupus nephritis: Isotype and antigen specificity make the difference. Autoimmunity reviews. 2015.
3. Pan XF, Gu JQ, Shan ZY. Patients with systemic lupus erythematosus have higher prevalence of thyroid autoantibodies: a systematic review and meta-analysis. PloS one. 2015;10(4):e0123291.
4. Ceccarelli F, Perricone C, Massaro L, Cipriano E, Alessandri C, Spinelli FR, et al. Assessment of disease activity in Systemic Lupus Erythematosus: Lights and shadows. Autoimmunity reviews. 2015.
5. Makashir SB, Kottyan LC, Weirauch MT. Meta-analysis of differential gene co-expression: application to lupus. Pacific Symposium on Biocomputing Pacific Symposium on Biocomputing. 2015:443-54.
6. Dema B, Charles N. Advances in mechanisms of systemic lupus erythematosus. Discovery medicine. 2014; 17(95):247-55.
7. Kunz M. Lupus erythematosus. Part I: epidemiology, genetics and immunology. Journal der Deutschen Dermatologischen Gesellschaft = Journal of the German Society of Dermatology : JDDG. 2013;11(8):709-19; quiz 20, -20; quiz 21.
8. Sinha R, Raut S. Pediatric lupus nephritis: Management update. World journal of nephrology. 2014;3 (2):16-23.
9. Rees F, Doherty M, Grainge M, Davenport G, Lanyon P, Zhang W. The incidence and prevalence of systemic lupus erythematosus in the UK, 1999-2012. Annals of the rheumatic diseases. 2014.
10. Ju JH, Yoon SH, Kang KY, Kim IJ, Kwok SK, Park SH, et al. Prevalence of systemic lupus erythematosus in South Korea: an administrative database study. Journal of epidemiology. Japan Epidemiological Association. 2014; 24(4):295-303.
11. Bronson PG, Komorowski LK, Ramsay PP, May SL, Noble J, Lane JA, et al. Analysis of maternal-offspring HLA compatibility, parent-of-origin effects, and noninherited maternal antigen effects for HLA-DRB1 in systemic lupus erythematosus. Arthritis and rheumatism. 2010; 62(6):1712-7.
12. Somers EC, Antonsen S, Pedersen L, Sorensen HT. Parental history of lupus and rheumatoid arthritis and risk in offspring in a nationwide cohort study: does sex matter? Annals of the rheumatic diseases. 2013; 72(4):525-9.
13. Tiffin N, Adeyemo A, Okpechi I. A diverse array of genetic factors contribute to the pathogenesis of systemic lupus erythematosus. Orphanet journal of rare diseases. 2013;8:2.
14. Deng Y, Tsao BP. Genetic susceptibility to systemic lupus erythematosus in the genomic era. Nature reviews Rheumatology. 2010;6(12):683-92.
15. Lewis CM. Genetic association studies: design, analysis and interpretation. Briefings in bioinformatics. 2002;3(2):146-53.
Campbell H, Rudan I. Interpretation of genetic association studies in complex disease. The pharmacogenomics journal. 2002;2(6):349-60.
17. Lawson HA, Cheverud JM, Wolf JB. Genomic imprinting and parent-of-origin effects on complex traits. Nature reviews Genetics. 2013;14(9):609-17.
18. Patten MM, Ross L, Curley JP, Queller DC, Bonduriansky R, Wolf JB. The evolution of genomic imprinting: theories, predictions and empirical tests. Heredity. 2014;113(2):119-28.
19. Guilmatre A, Sharp AJ. Parent of origin effects. Clinical genetics. 2012;81(3):201-9.
20. Wolf JB, Hager R, Cheverud JM. Genomic imprinting effects on complex traits: a phenotype-based perspective. Epigenetics : official journal of the DNA Methylation Society. 2008;3(6):295-9.
21. Miller SA, Dykes DD, Polesky HF. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic acids research. 1988;16(3):1215.
22. Howey R, Cordell HJ. PREMIM and EMIM: tools for estimation of maternal, imprinting and interaction effects using multinomial modelling. BMC bioinformatics. 2012;13:149.
23. Ainsworth HF, Unwin J, Jamison DL, Cordell HJ. Investigation of maternal effects, maternal-fetal interactions and parent-of-origin effects (imprinting), using mothers and their offspring. Genetic epidemiology. 2011;35(1):19-45.
24. Farid TM, Abd El Baky AM, Khalefa ES, Talaat AA, Mohamed AA, Gheita TA, et al. Association of tumor necrosis factor-alpha gene polymorphisms with juvenile systemic lupus erythematosus nephritis in a cohort of egyptian patients. Iranian journal of kidney diseases. 2011;5(6):392-7.
25. Mostowska A, Lianeri M, Wudarski M, Olesinska M, Jagodzinski PP. Vitamin D receptor gene BsmI, FokI, ApaI and TaqI polymorphisms and the risk of systemic lupus erythematosus. Molecular biology reports. 2013;40(2):803-10.
26. Sakulpipatsin W, Verasertniyom O, Nantiruj K, Totemchokchyakarn K, Lertsrisatit P, Janwityanujit S. Vitamin D receptor gene BsmI polymorphisms in Thai patients with systemic lupus erythematosus. Arthritis research & therapy. 2006;8(2):R48.
27. Namjou B, Kim-Howard X, Sun C, Adler A, Chung SA, Kaufman KM, et al. PTPN22 association in systemic lupus erythematosus (SLE) with respect to individual ancestry and clinical sub-phenotypes. PloS one. 2013;8(8):e69404.
28. Mandal M, Donnelly R, Elkabes S, Zhang P, Davini D, David BT, et al. Maternal immune stimulation during pregnancy shapes the immunological phenotype of offspring. Brain, behavior, and immunity. 2013;33:33- 45.
29. Zager A, Peron JP, Mennecier G, Rodrigues SC, Aloia TP, Palermo-Neto J. Maternal immune activation in late gestation increases neuroinflammation and aggravates experimental autoimmune encephalomyelitis in the offspring. Brain, behavior, and immunity. 2015;43:159-71.
30. Jaeggi E, Laskin C, Hamilton R, Kingdom J, Silverman E. The importance of the level of maternal anti-Ro/ SSA antibodies as a prognostic marker of the development of cardiac neonatal lupus erythematosus a prospective study of 186 antibody-exposed fetuses and infants. Journal of the American College of Cardiology. 2010;55(24):2778-84.
31. Vinet E, Pineau CA, Clarke AE, Fombonne E, Platt RW, Bernatsky S. Neurodevelopmental disorders in children born to mothers with systemic lupus erythematosus. Lupus. 2014;23(11):1099-104.
32. Picascia A, Grimaldi V, Pignalosa O, De Pascale MR, Schiano C, Napoli C. Epigenetic control of autoimmune diseases: From bench to bedside. Clinical immunology. 2015;157(1):1-15.
33. Luo S, Liu Y, Liang G, Zhao M, Wu H, Liang Y, et al. The role of microRNA-1246 in the regulation of B cell activation and the pathogenesis of systemic lupus erythematosus. Clinical epigenetics. 2015;7(1):24.
Recibido: Diciembre 10, 2015
Aceptado: Mayo 17, 2016
Correspondencia:
ggaravit@uninorte.edu.co
CLIC AQUÍ Y DÉJANOS TU COMENTARIO