La Investigación como Experiencia Pedagógica en Colombia
Martalucía Tamayo F.1,2, Nancy Gelvez1, Marcela Rodríguez1, Silvia Florez2,
Clara Varon3, Ma. Claudia Lattig1 y Jaime Bernal1
Resumen
Durante los pasados 4 años, realizamos un programa de tamizaje entre 1,763 individuos sordos a través del país, para detectar Síndrome de Waardenburg (WS), en el cual identificamos 140 individuos afectados pertenecientes a 95 familias.
Se confirmó el diagnostico clínico de WS en 95 propositus y en 45 parientes afectados, lo que significa una frecuencia del 5.38% de WS entre la población sorda Colombiana. Exámenes audiológicos, oftalmológicos y genéticos fueron practicados para confirmar el diagnostico.
Siguiendo la clasificación del consorcio del WS, modificada por varios autores, basados en el Índice de Waardenburg (WI) que define el tipo de WS, un índice >1.95 fue considerado WS1 (con dystopia canthorum) mientras que un índice por debajo de este valor fue WS2. En nuestra población el 62.1% de los pacientes fueron clasificados como WS2 y el 37.9% como WS1.
Presentamos los resultados del estudio de manifestaciones clínicas, analizando la presencia de estas, la severidad y simetría de los hallazgos clínicos en esta población afectada.
En total, entre los 95 propositus, encontramos que el hallazgo clínico más frecuente fue la sordera sensorial (100%), seguida de raíz nasal amplia (58.9%), un familiar de primer grado afectado (37.89%), heterocromia irides (36.8%), hipopigmentación de la piel (31.57%), mechón blanco de pelo (28.42%), iris azul intenso (27.36%), sinofris (12.63%), canas prematuras (10.52%), ptosis palpebral (9.47%) e hipoplasia del ala nasal (1.05%). (Lea también: Editorial: Propuesta de un Programa para Formación del Médico del Futuro)
Nuestros datos confirman una interesante variabilidad inter e intra familiar en cuanto a manifestaciones fenotípicas, así como una extrema variabilidad en la expresión de los genes comprometidos en esta enfermedad.
Las dificultades en la investigación, la practica tutorial de estudiantes y el proceso que deben seguir alumnos y maestros en esta clase de trabajos, demuestran una vez mas la importancia de utilizar la investigación científica como una experiencia pedagógica.
Palabras clave: Síndrome de Waardenburg, sordera, dominante, hipopigmentación, heterocromia iris, mutación, PAX3, MITF.
Introducción
El Síndrome de Waardenburg (WS) es un desorden autosómico dominante que se caracteriza por sordera e hipopigmentación en piel, ojos y cabello. Este síndrome es clínica y genéticamente heterogéneo, por lo cual ha sido subdividido en cuatro tipos.
Se han realizado diferentes estudios de ligamiento encontrando que el gen PAX 3 es el causante del WS tipo 1 y 3, el gen MITF es el causante del WS tipo 2 y el gen EDNRB o EDN 3 es el causante del WS tipo 4 (Winship y Beighton, 1992; Reynolds y col., 1995; Read y Newton, 1997).
En un estudio piloto, realizado por el programa de estudios genéticos de enfermedades visuales y auditivas del Instituto de Genética Humana en 1992 y de la Fundacion Oftalmologica Nacional, se logró establecer la causa de la sordera en 1715 individuos sordos de escuelas para niños con problemas auditivos, encontrando evidencia de causa ambiental en 579 (33.8%) de los casos, causa genética en 608 (35.4%); dentro de éstos últimos, 37 (2.16%) eran individuos con Síndrome de Waardenburg (Tabla 1) (Tamayo y col, 1992).
El Síndrome de Waardenburg es la primera causa de sordera dominante (Arias S. 1971; Martini Alessandro- Editor , 1996; Pardono et al, 2003). A nivel mundial se ha estimado que la frecuencia del síndrome en población sorda se encuentra entre el 2% y el 5% (Farrer y col, 1992; Toriello, 1995).
Recientemente, se decidió realizar un estudio completo sobre el síndrome de Waardenburg, con el fin de determinar la frecuencia exacta de éste en población sorda institucionalizada en Colombia (Tamayo et al, 1991). Además, identificar las mutaciones en los genes PAX3 y MITF causantes de este síndrome en nuestra población.
Marco teórico
Sindrome de Waardenburg (WS) (Waardenburg, 1951; Zlotorra et al, 1995)
• Hipoacusia NS, anomalías pigmentarias en ojo, piel y cabello.
• Clínicamente variable. Existen varios tipos: WS tipo 1, WS tipo2 (WS3, WS4).
• Genéticamente heterogéneo: Genes PAX3, MITF, (también implicados los genes EDN3, EDNRB, RET, SOX10).
• Herencia autosómica dominante. (Baldwin CT, et al, 1995; Bard LA. 1978; DiGeorge AM, 1960; Gorlin RJ. 1995). Criterios diagnósticos de WS: Criterios Mayores (Liu, Newton y Read AP, 1995):
• Hipoacusia neurosensorial
• Heterocromia del iris (parcial/ total). o iris azul intenso
• Poliosis
• Distopia cantorum
• Familiar afectado Criterios MENORES (McKusick, 1994; Partington, 1964; Pierpoint ,1993):
• Hipopigmentación en piel
• Sinofris
• Raíz nasal amplia
• Canas Prematuras
Índice de Waardenburg (WI)
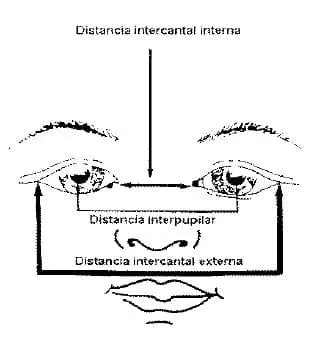
La distopia cantorum esta dada por el índice de Waardenburg (WI), el cual fue propuesto por Arias y Mota (Arias-Mota1978) (fig 1). Este índice es obtenido de la medida de la distancia cantal interna (a), la distancia Interpupilar (b) y la distancia cantal externa (c), donde se aplica la siguiente formula:
X = [2ª – (0.2119c + 3.909)]/c
Y = [2ª – (0.2497b + 3.909)]/b
W-Index = X + Y + a/b
Individuos con un valor del índice de Waardenburg mayor o igual a 1.95 son clasificados como WS tipo 1 y por debajo de ese valor, se clasifica WS tipo 2. (Hageman y Delleman, 1977; Farrer y col., 1994). IW ≥ 1.95 WS 1 (dystopia canthorum)-> GEN PAX3 IW GEN MITF
Genes responsables del síndrome de Waardenburg Asher y Friedman en 1990, sugirieron cuatro posibles localizaciones cromosomales en el humano, basados en la comparación con fenotipos mutantes en raton y hamster (Edery P, et al,. 1996; Hageman MJ., Delleman JW. 1977).
Los cuatro posibles loci fueron: en el brazo largo del cromosoma 2 (2q) cerca al gen de la fibronectina 1 (FN1); en el brazo corto del cromosoma 3 (3q) cerca al proto-oncogen RAF1; en el cromosoma 3q cerca al gen de la rodopsina (RHO) y en el cromosoma 4p cerca del gen KIT(Ommenn y McKusick,1979; Shah, 1981; Puffenberger, 1994 ).
Se han implicado los genes PAX3 (WS tipo 1 y 3), MITF (WS tipo 2) (Lalwani, Morell, Friedman, 1994) y más recientemente, los genes EDN3, EDNRB, RET y SOX10 (Ishikiriyama S. 1993; Klein D. 1950.
Objetivo general del trabajo
Definir las características fenotípicas del Síndrome de Waardenburg (WS), e identificar las mutaciones en los genes PAX3 y MITF causantes de esa enfermedad, en una población sorda institucionalizada en Colombia
Objetivos específicos del trabajo
• Determinar la frecuencia del síndrome de Waardenburg en la población sorda estudiada.
• Caracterizar las mutaciones presentes en los genes PAX3 y MITF.
• Establecer la correlación genotipo-fenotipo en los casos en que se encuentre mutación.
Materiales y Métodos
Población
Programa de tamizaje desarrollado durante 3 años para detectar WS. Se visitó cada una de las instituciones para sordos del país y a los individuos seleccionados se les realizó el diagnóstico clínico de WS de acuerdo al valor del índice de Waardenburg y a las características propias del síndrome.
Se completó un formato de historia clínica en cada uno de ellos, teniendo en cuenta los antecedentes familiares. Nuestro equipo médico realizó un completo examen genético y audiológico a cada uno de los individuos.
Con firma previa del consentimiento informado, se realizó toma de muestra de sangre periférica a cada uno de los individuos incluidos en el estudio.
En general, se identificaron 140 propósitos afectados con WS clásico y 45 familiares afectados. Para un total de 95 familias.
Técnicas moleculares
Secuenciación automática: Después de haber realizado SSCP, se procedió a la secuenciación. El DNA a ser secuenciado es obtenido mediante PCR (reacción en cadena de la polimerasa). El sistema automáticamente controla las condiciones de temperatura, tiempo y voltaje de la electroforesis.
Cuando cada ddNTP pasa por el laser una luz fluorescente es emitida al detector y los datos de cada muestra son inmediatamente almacenados y luego transferidos al software de análisis. Allí, cada secuencia es analizada y se determina la presencia de cualquier variante alélica.
1 INSTITUTO DE GENÉTICA HUMANA, Pontificia Universidad Javeriana.
2 FUNDACIÓN OFTALMOLÓGICA NACIONAL.
3 FUNDACIÓN OFTALMOLÓGICA DE SANTANDER – Foscal.
