Artículos Científicos
Académica Asociada Margaret Ordóñez-Smith de Danies*
Definición de la Resistencia
De acuerdo al CDC (Centro de Control de Enfermedades) de Estados Unidos, la resistencia se da cuando un medicamento deja de inhibir el crecimiento o matar a un microorganismo.
Hay dos tipos de resistencia: la adquirida y la clínica. La adquirida se realiza en el laboratorio a través de las mutaciones o por transferencia de genes, sistemas de conjugación, transducción y transformación(1).
La resistencia clínica es la observada en la práctica médica, donde no todos los organismos son igualmente, sensibles o igualmente resistentes a cierto antibiótico y que requiere la ayuda de técnicas de laboratorio para medir cuantitativamente o cualitativamente dicho proceso. (Lea también: Métodos Bacteriológicos para la Resistencia Bacteriana, Neumococos)
Mecanismos de la Resistencia Bacteriana
En la detección de la resistencia bacteriana se han identificado diferentes mecanismos utilizados por las bacterias: destrucción o inactivación enzimática, cambios en la permeabilidad de la membrana interna, alteraciones de los precursores de la pared celular, de la membrana y de los ribosomas. (2)
Para los años 90 se dispuso de una serie de antimicrobianos como betalactámicos, aminoglucósidos, macrólidos, sulfonamidas, trimetoprim, cloranfenicol, glicopéptidos, rifampicina, quinolonas, tetraciclinas (3).
Sabemos que la penicilina fue el primer antibiótico a partir del cual se desarrolló la clasificación de los betalactámicos tanto naturales (penicilina G) como semisintéticas de amplio espectro (ampicilina, amoxacilina, carbencilina).
Actualmente, podemos clasificar los betalactámicos en: A. Penicilinas: Meticilina, ampicilina, carbenicilinas, mezclocilina, piperacilina. B. Cefalosporinas: de primera generación (cefalexina, cefradina, cefalotina), de segunda generación (cefamandole, cefuroxima), de tercera generación (cefotaxima, ceftriaxona, ceftazidime) y cuarta generación (cefepime). C.
Cefamicinas: cefoxitina, cefotetan, cefmetazole. D.
Carbapenems: imipenem, meropenem. E. Monobatams: aztreonam. (4) Cuando la bacteria se vuelve resistente, produce enzimas, llamadas betalactamasas, las cuales se denominan por abreviaturas (tem-1, tem-2, ampC, ampR, ampG, ampD, OmpF, OmpC, etc).
Por ejemplo, las bacterias que producen tem-1 y tem-2 crean resistencia contra las cefalosporinas y penicilinas. Cuando producen tem-3 y tem-5, son resistentes a cefalosporinas, penicilinas y cefotaxima. Cuando tienen el oxa- 1 y PSE2 son resistentes a las penicilina y cloxacilina.
En la literatura se han descrito más de 250 de estas enzimas (4,5). Cuando producen resistencia contra los macrólidos, lincosamidas, las enzimas son denominadas MLSb, contra la eritromicina erm, contra la tetraciclina marRAB, DHSP contra las sulfonamidas, DNA girasa, gyrA, norA, ofxA, cfxA, contra las quinolonas, CAT contra el cloranfenicol, ant (2”) Ia, ant (3”) Ia, aac (3) Ia, aac (6’) Ia, genes que producen resistente a los aminoglucósidos.
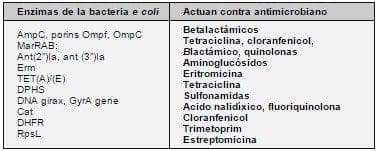
En la tabla 1 presentamos la Escherichia coli y las enzimas que producen resistencia bacteriana a los diversos antimicrobianos (6,7).
¿Cómo se Detecta la Resistencia Bacteriana en el Laboratorio?
Existen técnicas para saber como actúa un antimicrobiano ante una bacteria: in vivo (en el paciente) e in vitro (en el laboratorio). El médico utiliza un antibiótico adecuado gracias al antibiograma o prueba de susceptibilidad antimicrobiana reportado por el laboratorio.
El NCCLS (Comité Nacional de Control de Calidad de los Estándares) (8) tiene aprobada s 3 técnicas:
- MIC Concentración mínima inhibitoria (sistematizada).
- Difusión en disco.
- Test E.
Estas técnicas se utilizan para bacterias de crecimiento rápido (9). Para bacterias anaerobias se aplica la dilución en caldo o dilución en agar (10,11,12,13).
Sin embargo el test E está en estudio de aprobación para las bacterias anaerobias (14).
TABLA 1. ENZIMAS DE LA BACTERIA ESCHERICHIA COLI
Y SUS RESISTENCIAS A DIVERSOS ANTIBIÓTICOS
Detección de Betalactamasas
Varios científicos han descrito diferentes técnicas.
En 1972 Rosen inventó el método acidométrico, en 1975 Catlin usó el método iodométrico en papel impregnado con almidón y penicilina para ver el cambio de yodo a yoduro, en 1977 Slack usó el papel impregnado con penicilina y pH indicador, el cual al romper el anillo betalactámico forma el ácido penicilóico y en 1980 otros investigadores impregnaron sticks (palitos) con nitrocefina y una cefalosporina cromogénica.
Estos sticks viran a color rosado cuando hay producción de betalactamasa; se usan para Neisseria, Haemophilus, Staphylococcus o Bacteroides (3).
Detección de las Betalactamasas de Espectro Extendido
ESBLs: es una betalactamasa de espectro extendido, causada por mutación de plásmidos, tipo TEM, SHV, CTX-M, OXA y ya hay más de 150 enzimas conocidas. (1,4,7,15). La resistencia a cefalosporinas fue detectada por primera vez en Alemania en 1983, luego en una epidemia en Francia en 1985.
La ESBLs se ha reportado en bacterias como Klebsiella pneumoniae (16), Escherichia coli, Enterobacter, Salmonella, Proteus, Aeromonas y Pseudomonas. Se pueden detectar con dilución en caldo por MIC, sistema automatizado de Vitek o Microscan, test E o por el doble disco de diagnóstico.
El NCCLS recomienda para la Klebsiella y E coli hacer un primer descarte si los halos de inhibición del antibiograma (Tabla 2), luego sí proceder a realizar la técnica para detectar ESBLs (8,15).
Hay también resistencia a las betalactamasas en las bacterias anaerobias como Bacteroides thetaiotaomicron (17).
TABLA 2. MIC ZONA DE INHIBICIÓN PARA DETECTAR ESBLS EN K. PNEUMONIAE AND E.COLI

La técnica de ESBLs según el NCCLS debe ajustar el inóculo en un caldo a la concentración de la Escala de McFarland No. 5. Se inocula en agar Mueller Hinton y se colocan los discos de ceftazidime, cefotaxime con los combinados (18) (tabla 3):
TABLA 3. DISCOS COMBINADOS PARA DETECTAR LA ESBLS
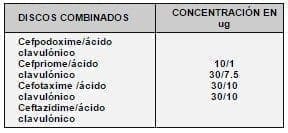
Cuando hay una diferencia mayor de 5 mm de los discos combinados con relación al halo de los sencillos se confirma la producción de ESBLs o con el procedimiento sistematizado cuando da >2mg/L. Por ejemplo, con el método de difusión en disco, si el combinado tiene un halo de inhibición de 20mm y el sencillo de 14 mm, quiere decir, que sí es productora de ESBLs.
El Centro de Control de Enfermedades de los Estados Unidos afirma además que la forma alargada de la cefalexina con el augmentin y la forma elíptica de la cefotaxima con el imipenem indican la presencia de la enzima ESBLs en la técnica de difusión en disco.
Así mismo, si el halo de inhibición de ampicilina es superior al diámetro de la cefotaxima, se detecta la cefalosporinasa (1).
Clases de los Staphylococcus
Hay varias clases de Staphylococcus aureus, que cada día presentan mayor resistencia. En 1944 había un 5% de resistencia a la penicilina, en 1949 un 50% y en 1995 un 90%(4). En la Tabla 4 se clasifican de acuerdo a la resistencia.
TABLA 4. CLASIFICACIÓN DEL STAPHYLOCOCCUS AUREUS
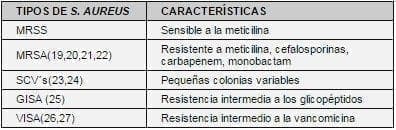
El MRSA (Staphylococcus aureus resistente a la meticilina) se puede detectar por diferentes técnicas: difusión en disco, aglutinación por latex o por Vitek (sistema automatizado) (19,20). El método de difusión en disco con agar Mueller Hinton se puede emplear para determinar el MRSA, con cefotaxima da <27mm, con moxalactam da o 20mm.
Cuando la oxacilina es >20mm, indica que el S aureus es sensible a la ampicilina, amoxacilina, amoxacilina/ acido clavulónico, o sublactam/ampicilina. En este año François (22) ha descrito otro sistema para determinar MRSA.
Recientemente se describió el medio de cultivo con agar manitol sal con cloruro de litio y azul de anilina y con cloruro de sodio al 5.5%, con suplementos de polimixina B y oxacilina para detectar S aureus resistente a oxacilina ( 28), e informar el resultado entre las 24 a 48 horas de la siembra.
Las colonias MRSA toman el color azul, se confirman con la coagulasa, catalasa, staphtect plus, dry spot o PBP2. Además se le hace control de calidad ya sea positivo para S aureus MRSA (ATCC 433000),o negativo para S aureus MSSA (ATCC 25923) o E coli ATCC 25922.
También existe el Staphylococcus coagulasa negativa que produce resistencia a la meticilina, se puede detectar por aglutinación de latex (29). Se han encontrado Staphylococcus epidermidis resistentes a la meticilina (21,30,31).
Enterococcus
Hay dos especies de Enterococcus, faecalis y faecium que son responsables de la mayoría de las bacteremias, infecciones en cirugía, infecciones de las vías urinarias y endocarditis en los Estados Unidos (32).
Están los HLR altamente resistentes a los aminoglucósidos y los VRE (Enterococcus resistentes a Vancomicina), descritos por primera vez en 1992 por el NCCLS. Existen varios fenotipos: van A: vancomicina y teicoplanina resistentes y van B: vancomicina resistente, pero sensible a la teicoplanina, y los van C-1 y van C-2/3 (33,34,35). Para ello hay dos técnicas: 1.
Difusión en disco con el agar Mueller Hinton, llevando el inóculo a la concentración de 0.5 de la Escala de McFarland, se deja por 18 horas con un disco de gentamicina de 120ug y estreptomicina de 300ug, la interpretación es: resistente con 6 mm, inconcluso 7 a 9 mm, sensible > o igual a 10 mm. 2.
El sistematizado MIC (concentración mínima inhibitoria) empleando 500 ug de gentamicina, 1000 ug de estreptomicina. Se deja 24 a 48 horas a temperatura ambiente y si crece es porque hay resistencia. El control de calidad se hace con Enterococcus faecalis ATCC 29212 con la gentamicina da halos entre 16 a 22 mm, y con la estreptomicina de 14 a 19 mm. (8). Con el Enterococcus feacalis ATCC 51299 ambos halos serían resistentes.
Hay limitaciones en el sistema automatizado Vitek GSP-418 (37).
Existe otro medio de cultivo para detectar directamente la bacteria VRE, que consiste en inocular la muestra en el medio y a las 24 a 48 horas la colonia toma el color negro, que confirma la resistencia a la vancomicina. Este medio tiene un suplemento de meropenem y vancomicina. Control de calidad con Enterococcus feacalis NCTC 12201 como control positivo y con E faecalis ATCC 33186 control negativo (36).
