Perspectiva del Diagnóstico y Tratamiento del Cáncer
Andrés F. Cardona1,2,3, Oscar Arrieta4, Zyania Lucía Zatarain-Barron4, Christian Rolfo5, Camila Ordoñez6, Alejandro Ruiz-Patiño1,3
Resumen
La comprensión de que la progresión del cáncer requería la interacción de múltiples genes proporcionó una de las razones fundamentales. Para embarcarse en 1986, en el proyecto genoma humano. Solo con una secuencia del genoma de referencia podría entenderse el espectro completo de cambios somáticos que conducen al cáncer.
Desde su finalización en 2003, la secuencia del genoma humano de referencia ha cumplido su promesa como herramienta fundamental para esclarecer la patogénesis de diversas neoplasias.
Los recientes avances biotecnológicos han llevado a la identificación de características biológicas complejas y únicas asociadas con la carcinogénesis. La perfilación del ADN tumoral libre circulante y de las células neoplásicas. Así como de factores relacionados con inmunidad, análisis de proteínas y del ARN, permiten optimizar el diagnóstico y tratamiento del cáncer.
En consecuencia, la búsqueda de respuestas con base en experimentos clínicos ha evolucionado. Ppasando de los estudios centrados en un tipo específico de tumor a una o muchas características genómicas, independientes de la histología, y con base en diseños innovadores y adaptativos.
A continuación, revisamos los hitos y conceptos históricos clave en la genómica del cáncer y algunos de los descubrimientos novedosos en esta área.
Palabras clave: Cáncer; genómica; medicina de precisión; biomarcador; ADN; mutación.
Cancer Genomics Evolution
Abstract
The realization that cancer progression required the interaction of multiple genes provided
one of key rationales, in 1986, for embarking on the human genome project. Only with a reference genome sequence could the full spectrum of somatic changes leading to cancer be understood.
Since its completion in 2003, the human reference genome sequence has fulfilled its promise as a foundational tool to illuminate the pathogenesis of diverse neoplasms. In recent years, biotechnological breakthroughs have led to identification of complex and unique biologic features associated with carcinogenesis.
Tumor and cell-free DNA profiling, immune markers, and proteic and RNA analyses are used to identify these characteristics for optimization of anticancer therapy in individual patients.
Consequently, clinical trials have evolved, shifting from tumor type-centered to gene-directed, histology-agnostic, with innovative adaptive design tailored to biomarker profiling with the goal to improve treatment outcomes. Herein, we review the key historical milestones and concepts in cancer genomics, and some of the novel discoveries in this area.
Keywords: Cancer; genomics; precision medicine; biomarker; DNA; mutation.
Introducción
En 1971, la clonación del ADN recombinante, la técnica más novedosa para la época, estaba todavía en su infancia y aún no era capaz de lidiar con la tarea de desentrañar toda la complejidad de la célula humana.
Sin embargo, no fue sino hasta 1985 cuando Dulbecco presentó la prueba de que las mutaciones en los genes podrían causar cáncer. Un hecho que hoy damos por sentado, brindando el valor de la secuencia genómica como base para comprender la enfermedad (1).
La discusión contemporánea sobre la generación de la secuencia humana completa representó un salto asombroso ya que, en ese momento, secuenciar un solo gen de aproximadamente 1 kilobase era digno de una tesis, y el genoma humano era 3 millones de veces más grande.
La estructura repetida del genoma estaba bien caracterizada y algunos también la consideraban un obstáculo insuperable para tal esfuerzo y una base para la resistencia temprana al proyecto (2).
A pesar de estos obstáculos, un esfuerzo internacional condujo a la finalización del genoma de referencia humano en 2003 (International Human Genome Sequencing Consortium 2004), y con esta reseña completa, fue posible probar el valor de la genómica para descifrar los cambios que conducen al cáncer. Los datos que han surgido desde 2003 respaldan de manera abrumadora el valor de esta visión y han cambiado la forma en que se investiga y se comprenden las neoplasias.
Período de secuenciación del genoma humano
Durante el período de secuenciación del genoma humano (1990-2003) (http://www.ornl.gov/sci/techresources/Human_Genome/project/about.shtml), la investigación continuó acumulando conocimiento sobre la clonación, permitiendo mejorar la capacidad de secuenciación lo que llevó a identificar la mayoría de los oncogenes y genes supresores de tumor.
Un inventario de las alteraciones asociadas al cáncer determinó la presencia de 291 genes conductores, lo que corresponde al ~1% de la secuencia codificante (3). Los primeros estudios en profundidad observaron que el 90% de estos genes presentaban mutaciones somáticas. Cerca del 20% tenía alteraciones germinales, y el 10% contenía ambas categorías.
Las formas más comunes de variación en el repertorio mutaciones contenido hasta 2004, fueron las traslocaciones que conducían a la generación de proteínas de fusión con carácter oncogénico. Hasta ese año, nadie había estudiado más de un puñado de genes tumorales, lo que representó el cambio más significativo en el conocimiento para la oncología actual.
Ese fue el estado de la investigación en el umbral de la “era genómica” del cáncer, un tiempo anunciado por la disponibilidad del genoma de referencia y la espectacular explosión de datos. Alimentado por la introducción de sistemas operativos para el control masivo de información y el desarrollo de instrumentos de secuenciación paralela más económicos. Este año es el vigésimo aniversario de la publicación de ese notable hito en la ciencia: la finalización del genoma humano de referencia.
En esta coyuntura, recapitulamos algunos de los hallazgos y desafíos clave que han surgido del análisis de la secuencia del genoma del cáncer.
Nomenclatura de las alteraciones genómicas
La genómica, en lo que respecta al cáncer, es el estudio de cómo las alteraciones en el código genético dan lugar a células anormales. Aunque ciertamente no está tan rígidamente instalado como antes, es importante considerar el dogma central de la biología molecular que describe el flujo jerárquico de información genética desde el ADN hasta la proteína.
La información genética codificada en el ADN se transcribe a ARN mensajero (ARNm) por la ARN polimerasa, y el ARNm se traduce posteriormente a la proteína a través de los ribosomas, lo que da como resultado una expresión fenotípica del gen subyacente.
Este proceso de transferencia de información del ADN a la proteína funcional se conoce como expresión génica. Casi el 90% del ADN genómico humano se transcribe en ARN; sin embargo, solo del 1% al 2% de las transcripciones se expresan como proteínas.
La mayoría de estas transcripciones son ARN no codificantes (ncRNA), que apenas estamos comenzando a reconocer por su importancia como reguladores de las funciones celulares.
Los errores en el procesamiento y transferencia de información genética conducen al desarrollo de mutaciones que dan lugar a una variabilidad significativa en el genoma pudiendo llegar a la génesis del cáncer (4).
El ADN es la unidad básica de información de todos los organismos.
Está compuesto de nucleótidos purina (adenina, guanina) y pirimidina (citosina, timina). Estos se organizan en secuencias para formar genes, que son las unidades estructurales y funcionales de información hereditaria dispuestas por combinaciones variadas de los cuatro nucleótidos.
El ADN se envuelve alrededor de las histonas y se agrupa como cromatina dentro del núcleo celular. Este material genético está organizado en cromosomas que se transportan por duplicado (diploide) con la excepción de los cromosomas sexuales, en la combinación XY.
Estas dos versiones separadas del mismo gen en las células diploides se conocen como alelos, que se encuentran en forma homocigótica (dos alelos idénticos) o heterocigótica (dos alelos diferentes). Los cambios en esta secuencia de ADN afectan potencialmente la función y el desarrollo de un organismo (5).
Las mutaciones son omnipresentes en las células neoplásicas (6). Estos cambios en la secuencia de ADN pueden conducir a una expresión o función proteica alterada. Hay múltiples tipos que pueden estar relacionadas con el desarrollo del cáncer. Y estas ocurren a través de varios mecanismos, que incluyen agresiones extrínsecas y defectos intrínsecos.
Las agresiones extrínsecas o mutágenos, son agentes naturales o artificiales que producen alteraciones en la secuencia del ADN. Estos mutágenos pueden ser biológicos (por ejemplo, virus de Epstein-Barr o el virus del papiloma humano), químicos (por ejemplo, bromuro de etidio, etc.) o radiológicos (por ejemplo, radiación ultravioleta [UV], radiación ionizante).
Los defectos intrínsecos incluyen fallas en la replicación o reparación del ADN. Dependiendo del tipo de células en las que ocurren estas mutaciones. Se clasifican en línea germinal (heredables y en células reproductoras) o somáticas (no heredables y no reproductivas).
La acumulación de mutaciones es un factor clave y responsable del desarrollo, crecimiento, metástasis y resistencia al tratamiento del cáncer (4).
Las mutaciones a menudo se clasifican según el grado de alteración del ADN.
Por ejemplo, las mutaciones puntuales o pequeños cambios suelen afectar a un solo gen, mientras que las anomalías cromosómicas alteran grandes segmentos del ADN.
Las mutaciones puntuales son el resultado de sustituciones, inserciones o deleciones de un solo par de bases. Las sustituciones de bases pueden provocar transiciones o transversiones.
Se produce una transición cuando una pirimidina se cambia por una pirimidina diferente o una purina se cambia por una purina alternativa después de una sustitución de base. Y una transversión, ocurre cuando una pirimidina se convierte en una purina o viceversa.
Si estas alteraciones resultan en la sustitución de un aminoácido diferente al esperado en una secuencia codificante para una proteína. Se conoce como una mutación de cambio de sentido o missense como es conocida en inglés.
Las inserciones o deleciones (indels) pueden dar como resultado desplazamiento en el marco en las que la lectura se desplaza de modo que se altera la secuencia de aminoácidos que se está codificando (frameshift).
Si el marco de codificación de aminoácidos es alterado por cualquier mutación de una manera que conduce a la introducción prematura de un codón de terminación (parada), resulta en una proteína truncada. Estas mutaciones, al igual que las puntuales que ocasionan el mismo resultado se conocen como mutaciones sin sentido (non-sense).
Las grandes alteraciones del ADN, como las anomalías cromosómicas o rearreglos, modifican la estructura de los cromosomas a través de la pérdida, la ganancia o el reordenamiento de segmentos.
Estas anomalías ocurren cuando los cromosomas sufren procesos de ruptura y reunión, lo que resulta en el intercambio o la pérdida de material genético dentro del mismo o entre diferentes cromosomas (Figura 1).
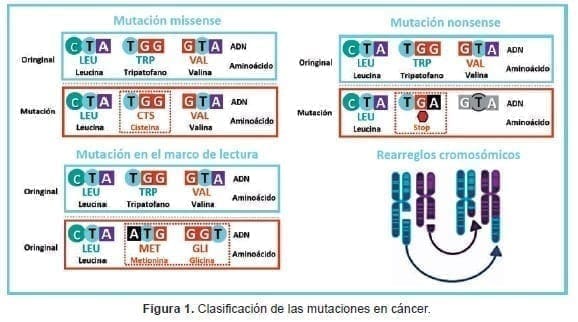
Si bien algunas anomalías cromosómicas son hereditarias, muchas ocurren de manera espontánea o como consecuencia de factores ambientales como la exposición a medicamentos, rayos X, rayos UV, u otros factores carcinogénicos. Hay cuatro rearreglos estructurales cromosómicos comunes que incluyen las inversiones, deleciones, duplicaciones y translocaciones (4).
Las inversiones ocurren cuando el segmento de un cromosoma se desprende y luego se vuelve a unir en la orientación inversa al mismo cromosoma (7).
Como sugiere el nombre, una deleción es la pérdida de un segmento cromosómico (como en el síndrome de Cridu- chat) de manera que el material genético perdido no se puede recuperar.
La duplicación de un segmento cromosómico puede ocurrir cuando una parte del cromosoma se copia y se inserta junto al segmento original en tándem o en orientación inversa (7).
Las translocaciones implican la transferencia de un segmento de un cromosoma a una parte diferente del mismo cromosoma o a un cromosoma completamente diferente.
Genéticamente, sobre todo si los puntos donde se da la ruptura o la reunión comprometen regiones críticas, puede dar como resultado el cambio de secuencia, posición y orden de algunos genes, lo que a menudo en cáncer, resulta en varias anomalías funcionales que implican una consecuencia biológica.
La leucemia mielógena crónica (LMC), una de las neoplasias relacionadas con una translocación mejor caracterizadas. Se inicia cuando la porción del cromosoma 9 que alberga el código genético de la tirosina-quinasa, el gen de la leucemia murina de Abelson (ABL), se une al gen de la proteína de la región de clúster de punto de ruptura (BCR) ubicada en el cromosoma 22. Formando el gen de fusión BCR-ABL, conocido como cromosoma Filadelfia.
El producto proteico de este rearreglo, exhibe una actividad tirosina-quinasa patológicamente incrementada, fomentando la proliferación celular. Conociendo este mecanismo esta translocación se convirtió en el objetivo de algunas de las primeras terapias contra el cáncer dirigidas molecularmente (8).
Además de las variaciones estructurales, también se producen aberraciones numéricas de los cromosomas que se denominan aneuploidía (una pérdida o una ganancia de uno o más cromosomas) y polisomías (uno o más genomas completos en una célula que pueden ser idénticos o distintos entre sí) (7).
Estos se han asociado con el desarrollo de algunas neoplasias como el neuroblastoma, los astrocitomas pediátricos y los condrosarcomas (9-11).
Modificaciones epigéneticas
El término epigenética denota cambios en el fenotipo de una célula sin afectar el genotipo, o la secuencia de la misma (12). Más recientemente, la epigenética se ha definido como todos los cambios hereditarios en la expresión génica y en la estructura de la cromatina que no están codificados en la secuencia propia del ADN (5).
En todos los organismos multicelulares eucariotas, los procesos de diferenciación están controlados a través de estos mecanismos, con la excepción de la maduración de las células T y B (5).
La regulación epigenética de los fenotipos incluye la metilación del ADN, las modificaciones de histonas y la expresión de micro-ARN (miARN). Los cambios en cualquiera de estos reguladores epigenéticos pueden provocar una alteración de la expresión génica y el desarrollo de cáncer y otras enfermedades crónicas (5).
La metilación del ADN:
Es la adición de un grupo metilo a las citosinas ubicadas en secuencias de ADN conocidas como islas CpG (repeticiones de citosina seguida de guanina). Es una de las primeras modificaciones epigenéticas descritas, y aproximadamente del 70% al 80% de las citosinas en estas estructuras están metiladas en células somáticas humanas (13). La adición de grupos metilo está mediada por un complejo de enzimas catalíticas conocidas como ADN metiltransferasas (DNMT).
Para contrarrestar la acción de las DNMT, las demetilasas eliminan estos grupos metilo, resultando en un balance homeostático de activación e inactivación de transcripción. Es entonces que los procesos de metilación del ADN juegan un papel crucial en el mantenimiento de la estabilidad del genoma, la inactivación del cromosoma X femenino. Así como en la regulación de la expresión génica y los procesos de desarrollo (14,15).
Las alteraciones en estos patrones fueron las primeras alteraciones epigenéticas identificadas en el cáncer. En las neoplasias se han identificado fenómenos de hipometilación, una disminución general de los patrones de metilación del ADN, así como la hipermetilación, un aumento global de la metilación de las islas CpG (15). Un ejemplo del segundo se ha identificado en los promotores del gen CDKN2A. El cual lleva a la pérdida de la expresión de los supresores tumorales p16 y p14arf y por ende. Promueve el proceso oncogénico por pérdida de tan importantes reguladores del ciclo celular (16).
Alternativamente, la hipometilación se ha identificado especialmente a nivel de los promotores de protooncogenes y factores de crecimiento. Fomentando la proliferación e inmortalización de células malignas (15).
Otro cambio epigenético importante que puede conducir al cáncer implica la modificación postraduccional de las histonas.
Vale la pena mencionar, que, al contener la cadena de ADN empaquetada, los cambios que involucren especialmente residuos que interactúan por diversas fuerzas electroquímicas sobre la misma, van a repercutir directamente en esta relación, fomentando o inhibiendo la condensación de la hebra. Los principales cambios sobre estas moléculas son la metilación, acetilación, fosforilación, ubiquitilación y sumoilación.
La metilación de regula varios procesos celulares como el procesamiento del ARN, la biosíntesis de ribosomas, la reparación del ADN, la transducción de señales y la regulación transcripcional. Se sabe adicionalmente que la metilación sobre residuos lisina/arginina es directamente responsable de la activación o represión transcripcional (5).
La acetilación y deacetilación, que están mediadas por enzimas conocidas como histonas acetiltransferasas (HAT) e histonas deacetilasas (HDAC), respectivamente, son fundamentales para regular la dinámica de la cromatina, la transcripción, el silenciamiento de genes, la progresión del ciclo celular, la apoptosis, la diferenciación, la replicación del ADN y la reparación del mismo.
Un de-sequilibrio en la ponderación de la acetilación y desacetilación de histonas está directamente relacionado con la tumorigénesis y la progresión de la enfermedad. Se han observado alteraciones de los genes que codifican las enzimas HAT y HDAC en leucemias y cánceres de colon, útero y pulmón. Y se han desarrollado de este proceso para atacar estas aberraciones en las células cancerosas (12).
La fosforilación de histonas es menos común en comparación con la acetilación y metilación. Sin embargo, este proceso sobre un residuo de serina inicia la respuesta al daño del ADN y sobre una treonina controla la estructura de la cromatina mediante la señalización de las proteínas modificadoras de marcas epigenéticas (12).
Los ncARN, como el miARN y el ARN de interferencia corta (siARN), se han identificado como reguladores clave adicionales de la expresión génica.
Al interferir con procesos como la traducción de ARNm, la metilación del ADN y las modificaciones de histonas, los miARN regulan procesos celulares como el desarrollo, la proliferación, la diferenciación y la muerte celular.
Estos miARN están constituidos por 20 a 22 nucleótidos de longitud y están ubicados en sitios frágiles que son susceptibles a amplificaciones, o ganancia en el número de copias.
Diferentes estudios han mostrado evidentes diferencias en los perfiles de expresión de miARN en tejidos normales frente a los tumorales. Adicionalmente, estos afectan a los genes supresores tumorales, los oncogenes y los genes de reparación del ADN (17).
Datos recientes han demostrado una utilidad emergente de los miARN como herramientas en el diagnóstico, pronóstico y tratamiento del cáncer (18). Pero aún queda mucho por dilucidar con respecto a la diversidad de funciones que desempeñan estos reguladores epigenéticos.
(Lea También: Métodos Básicos para Genotipificación en Cáncer)
Mecanismos de reparación del ADN
Las células son desafiadas constantemente por agresiones al genoma que podrían resultar en mutaciones. Existen varias vías de reparación y respuesta al daño del ADN para reconocer estos efectos relativamente comunes, y mantener la estabilidad e integridad genómica. Cuando estos procesos fallan es que puede originarse las diferentes alteraciones mencionadas previamente, iniciando el proceso oncogénico.
Dentro de los mecanismos involucrados en este proceso están la reparación por escisión de bases, la reparación por escisión de nucleótidos, la reparación por desajustes (MMR), y la reparación por escisión bicatenaria (19).
El primero hace referencia a la corrección de una o dos bases las cuales se encuentran alteradas. Generalmente por cambios químicos tales como oxidaciones que a su vez no modifican la estructura helicoidal de la cadena. Este mecanismo retira la base afectada al igual que un pequeño fragmento adyacente el cual es reparado por medio de una polimerasa.
En caso de que haya distorsión de la estructura de la hélice, ya sea por radiación ultraviolenta, mutágenos ambientales o exposición a agentes quimioterapéuticos, a menudo se elimina mediante reparación por la vía NER o de escisión de nucleótidos.
Los defectos en este mecanismo de reparación se asocian con envejecimiento prematuro y el cáncer. NER consiste en el reconocimiento del daño, el desenrollamiento del ADN alrededor de la lesión. La escisión y la modificación de la hebra alterada para generar la síntesis del nuevo ADN incluido con una ligadura final simultánea (20).
La reparación por la vía NER defectuosa:
Relacionada con el xeroderma pigmentoso (XP) da como resultado la presentación de múltiples cánceres de piel inducidos por rayos UV (20). Esta es una rara enfermedad hereditaria con carácter autosómico recesivo en la que el afectado muestra una marcada tendencia a desarrollar lesiones neoplásicas cutáneas dependientes de la exposición al sol. Los heterocigotos son frecuentemente asintomáticos y no desarrollan la enfermedad.
La lesión más significativa que produce la luz ultravioleta sobre el ADN consiste en la formación de dímeros de timina. Es decir, dos timinas adyacentes (en una misma cadena de ADN) las cuales se unen covalentemente, causando alteraciones en el proceso de replicación del ADN.
En humanos, varios complejos enzimáticos (fotoliasas y su cofactor FADH2) se hallan implicados en la reparación de este fenómeno por medio de un complejo enzimático que absorbe la luz y utiliza esta energía para romper el enlace que une el dímero, separando las dos timinas. Es entonces que esta enfermedad es consecuencia de la mutación de cualquiera de los siete genes implicados en este mecanismo de reparación (21).
El sistema MMR (del inglés mismatch repair)
Mantiene la estabilidad genómica corrigiendo las bases mal emparejadas formadas debido a la mutación, sustitución, deleción o inserción de nucleótidos. El MMR ocurre durante la fase S del ciclo celular en donde se da una corrección por un proceso de escisión de la base anómala e inserción de la correcta.
Algunas de las proteínas clave necesarias para el correcto funcionamiento del sistema MMR son MSH2, MLH1 y ADN polimerasas δ y ε. Las mutaciones en MSH2 y MLH1 están relacionadas con un fenotipo hipermutado y con la inestabilidad del genoma, evento relativamente frecuente en el cáncer de colon (22).
Durante una fase de replicación eficaz, las ADN polimerasas δ y ε son necesarias para la actividad de corrección. Y, si se produce una mutación en estas enzimas, los desajustes de las bases aumentan, dando lugar a mutaciones complejas (23,24). La pérdida de la función de la vía de reparación MMR está relacionada con cánceres esporádicos y hereditarios (25).
Las roturas de doble cadena del ADN son lesiones considerablemente lesivas y están relacionadas con la pérdida de grandes regiones cromosómicas, lo que conduce a un desgaste de la integridad genómica.
La falta de reparación de las roturas de doble hebra a menudo conduce a la pérdida de información genética, inestabilidad y fragmentación del genoma, reordenamiento cromosómico y muerte celular (25).
Una célula de mamífero emplea dos vías diferentes para restaurar las rupturas bicatenarias: recombinación homóloga y unión de extremos no homólogos. La recombinación homóloga requiere homología de la secuencia de ADN para reparar la lesión de ADN y está activa en las fases media S y G2 del ciclo celular.
Los cánceres hereditarios de seno y ovario están asociados con mutaciones en los genes BRCA1 o BRCA2. Ambas proteínas BRCA son necesarias para la reparación de roturas bicatenarias del ADN competente mediante recombinación homóloga.
La unión o reparación de extremos no homólogos no requiere paralelismo de secuencia para reparar roturas de doble cadena y es activa durante todo el ciclo celular. Pero es dominante en las fases G0/G1 y G2 (24,25).
Además de los mecanismos de reparación mencionados existe una serie de componentes que revierten directamente algunas lesiones del ADN en un proceso de un solo paso denominado reversión directa.
Por ejemplo, el daño inducido por los rayos UV se repara mediante enzimas conocidas como fotoliasas. Las lesiones oxidativas son reparadas directamente por la enzima metilguanina-metiltransferasa (MGMT) (24).
Los datos clínicos emergentes proporcionan evidencia de que la sobreexpresión de los factores de reparación del ADN puede tener un significado pronóstico y predictivo en diversas neoplasias, y la inhibición de la reparación del ADN se ha convertido en un objetivo prometedor para el cáncer de seno, próstata y ovario.
Autores
1 Andrés F. Cardona. MD, MSc, Alejandro Ruiz-Patiño.PhD. Fundación para la Investigación Clínica y Molecular Aplicada del Cáncer – FICMAC, Bogotá, Colombia.
2 Andrés F. Cardona. Grupo Oncología Clínica y Traslacional, Clínica del Country, Bogotá, Colombia.
3 Andrés F. Cardona, Alejandro Ruiz-Patiño. Grupo de Investigación en Oncología Molecular y Sistemas Biológicos (FoxG), Universidad El Bosque, Bogotá, Colombia.
4 Oscar Arrieta, Zyania Lucía Zatarain-Barron. Sección Oncología Torácica y Laboratorio de Medicina Personalizada del Cáncer, Instituto Nacional de Cancerología – INCaN, Ciudad de México, México.
5 Christian Rolfo. Departamento Oncología Clínica, Marlene and Stewart Greenebaum Comprehensive Cancer Center, Facultad de Medicina Universidad de Maryland, Baltimore, Maryland, Estados Unidos.
6 Camila Ordoñez. MD. Fundación para la Investigación Clínica y Molecular Aplicada del Cáncer – FICMAC, Bogotá, Colombia.