Anticuerpos anti-PDI disminuyen la Infección por Rotavirus
Para confirmar la especificidad del bloqueo de la actividad disulfuro isomerasa con los inhibidores químicos, se realizó la infección de las células en presencia de anticuerpos contra la proteína PDI.
A una concentración de 10 μg/ml de anticuerpo anti-PDI, la infección rotaviral disminuyó de manera dosis dependiente y la inhibición máxima fue del 90% (Fig. 4A).
El anticuerpo de conejo usado como control de isotipos no tuvo incidencia alguna sobre la infección, descartando cualquier interacción inespecífica de la región Fc del anticuerpo. (Lea también: Interferencia de la Infección por Rotavirus, Discusión)
De otro lado, al preincubar las células con anticuerpos anti-PDI, y luego adicionar el virus a 4°C por 1 hora, no se detectaron diferencias del virus adherido con respecto a las células sin tratamiento con anticuerpo (Fig.4B). Estos resultados reafirman que la PDI está implicada en un evento posterior a la unión del virus a la célula.
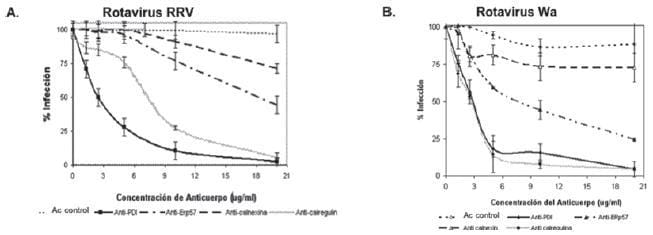
La interacción de las células con los anticuerpos anti-ERp57 (miembro de la familia de las PDIs) afectó la infección con rotavirus RRV en cerca del 50% solo a concentraciones relativamente altas de anticuerpo (18 μg/ml) y con un perfil lineal sugestivo de una reacción inespecífica (Fig. 5A).
Anticuerpos contra calexina (“chaperona” que actúa en el RE) presentaron un perfil similar de inespecificidad, pero con menor capacidad inhibitoria que para el caso de los anticuerpos dirigidos contra ERp57 (Fig. 5A).
Los anticuerpos anti-calregulina mostraron un efecto similar al de los anticuerpos anti-PDI pero con una afinidad promedio tres veces menor (Fig. 5A).
En el caso de la cepa Wa, los perfiles de inhibición producidos por los anticuerpos ensayados fueron similares a los descritos para la cepa RRV, excepto que los anticuerpos anti-calregulina produjeron un efecto indistinguible de aquel presentado por los anticuerpos anti-PDI (Fig. 5B).
El virus unido se cuantificó mediante ELISA de captura, capturando los antígenos con anticuerpo anti-rotavirus hecho en cabra y detectando con anti-rotavirus hecho en conejo con el sistema peroxidasa-OPD. El 100% de unión delta de absorbancia corresponde al delta de absorbancia de la sustracción de la absorbancia de células lisadas en ausencia de inhibidor y células sin virus.
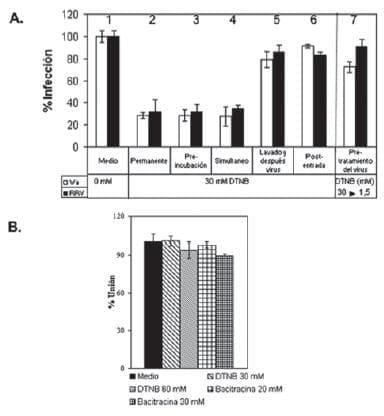
Figura 3. Efecto del DTNB en las diferentes etapas del proceso infectivo del rotavirus RRV y Wa en células MA104. En A, inmunocitoquimica de células infectadas con rotavirus Wa y RRV. (1) Infección sin inhibidor; (2) El proceso de infección se incubó permanentemente con DTNB; (3) se pre-incubó las células con DTNB por 30 minutos antes del reto; (4) se adicionó el virus y el DTNB simultáneamente, (5) se pre-incubó las células con el DTNB por 30 minutos y se retiró por lavados, después las células fueron retadas con el virus; (6) el DTNB se adicionó 1 h después de iniciado el proceso de internalización o post-adherencia y (7) el virus concentrado fue pretratado con 30 mM de DTNB, luego diluido hasta la concentración de trabajo y el DTNB hasta 1.5 mM. En B, unión a 4°C de rotavirus RRV a las células en presencia y ausencia de DTNB o bacitracina.
La proteína disulfuro isomerasa (PDI) se encuentra en la membrana de células permisivas a la infección por rotavirus, asociada a los microdominios lípidicos “rafts”.
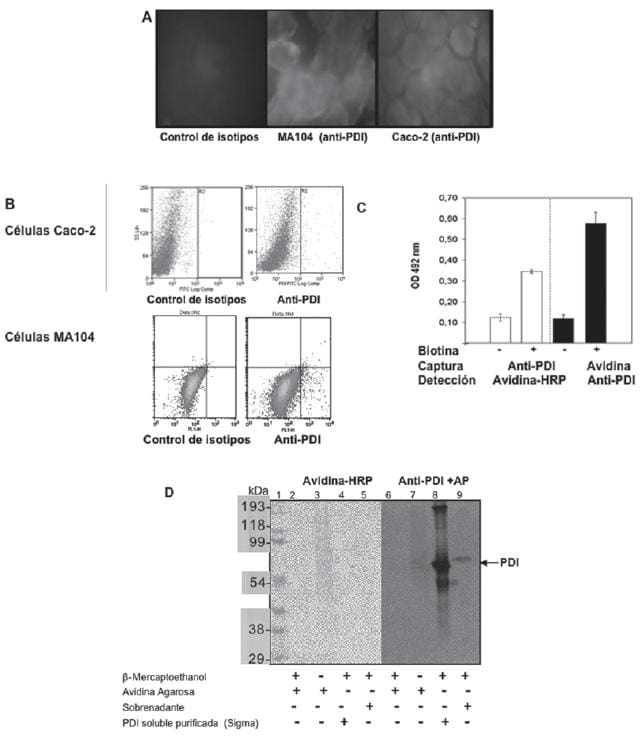
El carácter de proteína de superficie de la PDI en las células MA104 y Caco-2 fue analizada por inmunofluorescencia sobre monocapas celulares no permeabilizadas, señalando un perfi l positivo aunque de baja intensidad (Fig .6A).
La citometría de flujo de células en suspensión refleja que solo el 1.7% de la población presenta reactividad hacia el anticuerpo monoclonal anti-PDI (Fig. 6B). Para confirmar la presencia de PDI asociada a la membrana celular se marcaron las proteínas superficiales con el reactivo sulfo-NHS-SS-biotin, que adiciona reversiblemente una molécula de biotina a los grupos amino libres de las proteínas, en este caso a las proteínas asociadas a la membrana celular.
El lisado celular fue analizado mediante ELISA de captura usando alternativamente anticuerpos anti-PDI generados en cabra y la proteína avidina; para la detección se utilizó avidina-HRP, y anti-PDI generado en conejo, respectivamente.
Este ensayo mostró que el lisado celular contiene PDI biotinilada que está presente en las proteínas asociadas a la membrana celular accesibles de ser marcadas (Fig 6C). Al lisado celular se le adicionó Avidina-agarosa con el objeto de separar las proteínas que incorporaron el sulfo-NHS-SS-biotin.
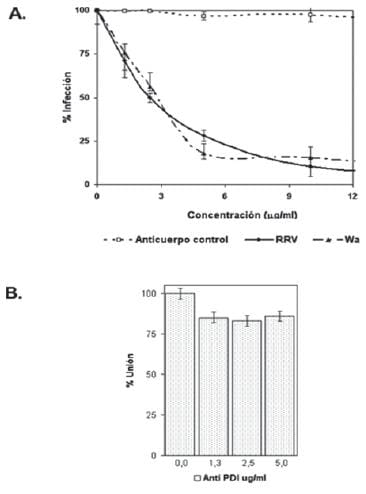
Figura 4. Disminución en la infección por rotavirus RRV y Wa en células MA104 con Anti-PDI en un paso post-unión. En A, las células se incubaron en ausencia y presencia del anticuerpo durante 30 minutos y luego se desafi aron con el rotavirus, la infección se dejó por 12h. El número de UFF se determinaron mediante inmunocitoquimica. En B, Unión a 4°C de rotavirus RRV a las células en presencia y ausencia del Anti-PDI. El virus unido se cuantifi có mediante ELISA de captura, capturando los antígenos con anticuerpo anti-rotavirus hecho en cabra y detectando con anti-rotavirus hecho en conejo con el sistema peroxidasa-OPD. El 100% de unión delta de absorbancia corresponde al delta de absorbancia de la sustracción de la absorbancia de células lisadas en ausencia de inhibidor y células sin virus.
Figura5. Disminución en la infección por rotavirus RRV (A) y Wa (B) en células MA104 con anticuerpos dirigidos contra diferentes chaperonas. Las células se incubaron en ausencia y presencia del anticuerpo durante 30 minutos y luego se desafi aron con el rotavirus, .la infección se dejó por 12h. El número de UFF se determinaron mediante inmunocitoquimica con el sistema peroxidasa-aminocarbazo.
Figura 6. Detección de PDI en la membrana celular. A. Inmunofl uorescencia indirecta no permeable sobre células MA104 y Caco-2, usando anticuerpo monoclonal anti-PDI a 10 μg/ml y anti-ratón FITC 0,7 μg/ml. B. Citometria de Flujo, marcaje de las células Caco-2 y MA104 en suspensión, con anticuerpo primario anti-PDI a 50 ug/ml, anticuerpo secundario anti-ratón FITC 0,7 ug/mL. C y D Biotinilación reversible de las proteínas superficiales de células MA104; las células fueron marcadas con el reativo sulfo-NHS-SS-Biotin y lisadas.
Una fracción del lisado celular biotinilado fue analizado por ELISA de captura utilizando alternativamente anticuerpos anti-PDI generados en conejo o proteína avidina para capturar los antígenos, la detección de éstos se hizo con avidina-HRP y anticuerpos anti-PDI respectivamente. C.
Las proteínas biotiniladas de la otra fracción del lisado celular se aislaron con agarosa-avidina. Las proteínas inmovilizadas y aquellas del sobrenadante se separaron mediante electroforesis denaturante bajo condiciones reductoras y no reductoras. El corrido electroforético se transfirió a una membrana de PVDF y el Western blot se analizó con avidina-HRP y anticuerpos anti-PDI.
Se detectó un perfil continuo de proteínas que reaccionan con avidina-HRP en las muestras que contenían las proteínas celulares marcadas con biotina e inmovilizadas con avidina-agarosa, cuando éstas fueron analizadas en ausencia del reductor 2-ME (Fig. 6D).
Este patrón de proteínas biotiniladas se perdió cuando las proteínas inmovilizadas se trataron con 2-ME (Fig. 6D), indicando que la unión de la biotina ha sido revertida por la acción del reductor. Cuando se analiza la fracción proteica del lisado no inmovilizado (sobrenadante), no se detectó interacción con avidina-HRP, indicando la ausencia de biotina en las proteínas analizadas (Fig. 6D).
Esta misma membrana del “Western blotting” se analizó con anticuerpos anti-PDI y el sistema fosfatasa alcalina (AP); se observaron bandas de interacción a la altura del peso molecular de la PDI mostrando las formas reducida y oxidada de la proteína (Fig. 6D). En el sobrenadante no biotinilado, que contiene la fracción citoplasmática, el anticuerpo anti-PDI detectó una banda mas intensa respecto a la banda separada con avidina (Fig. 6D).
Estos resultados junto con los encontrados por las técnicas de citometría de flujo e inmunofluorescencia concuerdan con lo ya reportado acerca de la baja concentración de PDI en la membrana celular.
Se ha reportado que proteínas receptoras para rotavirus, como la Hsc70 y la integrina 3, se encuentran en los microdominios lipidicos “rafts” (Pavel et al., 2004), por ello se decidió explorar la posibilidad de que PDI estuviera asociada a los “rafts”.
Para esto las fracciones de lisados celulares enriquecidas en membranas, separadas por gradientes de sacarosa y tratadas, como se describió en Materiales y Métodos para deshacer los “rafts”, fueron analizadas por “Western blotting” y ELISA de captura.
Mediante “Western blotting” se encontró a PDI y la proteína Hsc70 en las fracciones de menor densidad, correspondientes a aquellas enriquecidas en membranas que incluyen los microdominos lipídicos “rafts”; también se detectaron estas proteínas en las fracciones del extremo de alta densidad, que corresponde a las proteínas citoplasmáticas solubles (Fig. 7A).
Cuando las células se trataron con -metilciclodextrina antes de ser lisadas y separadas en el gradiente, no se detectaron PDI y Hsc70 en las fracciones de baja densidad, lo cual es compatible con la disociación de los “rafts” ocasionada por el tratamiento previo con -metilciclodextrina (Fig. 7B).
Para determinar si en estas condiciones experimentales PDI está unida por interacción proteínaproteína a moléculas receptoras como la Hsc70 y la v3, los lípidos de cada fracción del gradiente de sacarosa fueron solubilizados, incubando cada fracción con octilglucósido, Triton al 1% y -ciclodextrina.
Posteriormente, cada fracción se sometió a inmunoprecipitación con anti-PDI; se encontró que PDI co-inmunoprecipita con Hsc70 y la integrina 3 en las fracciones del gradiente analizadas (Fig. 7C).
Estos resultados se confi rmaron mediante ELISA de captura, determinándose nuevamente interacciones que implicaban PDI, Hsc70 e integrina 3 (Fig. 7D). El reconocimiento de los anticuerpos específicos anti-ERp57 no presentó afinidad significativa por los componentes del complejo examinado.
En conjunto, los resultados obtenidos por las diferentes técnicas de inmunodetección y marcaje, indican la presencia en muy baja concentración de la proteína PDI asociada a la membrana celular en células MA104 y Caco-2; haciendo parte de los microdominios lípidicos “rafts” y formando complejos con las proteínas Hsc70 e integrina 3, utilizadas como receptores por las proteínas VP7 y VP4 del rotavirus en el proceso de internalización.
CLIC AQUÍ Y DÉJANOS TU COMENTARIO