Con el fin de obtener una secuencia de DNA que represente el codificado de una proteína particular, el enfoque ideal es comenzar con su correspondiente mRNA el cual después de todo, es el molde empleado para la traducción en la proteína in vivo. La existencia de la transcripción reversa (por la transcriptasa reversa AMV, del virus de la mieloblastosis aviar, enzima que copia una cadena de DNA a partir de un molde de RNA), es una opción que se emplea para resolver esta clase de problemas en la clonación de secuencias de DNA. La transcripción reversa es extremadamente fácil para mRNAs que posean colas de poli (A) en su extremo 3’ (al igual que otras enzimas que sintetizan DNA, la transcriptasa reversa no puede iniciar la formación de una cadena polinucleotídica sin un extremo cebador) (figura 7).
El producto de reacción de la transcriptasa reversa, es una molécula de DNA de doble cadena complementaria al mRNA que se utilizó como molde. El único problema práctico es la tendencia que tiene la transcriptasa reversa in vitro a detenerse en varios puntos antes de alcanzar el extremo 5’. Otra característica importante de esta enzima es que cuando alcanza el terminal 5’, se desliza de 10-20 bp por fuera del molde, generando un gancho. Este puede ser empleado para iniciar la síntesis de la banda de DNA complementaria.
En esta unión el RNA se degrada por tratamiento con álcali, para producir un DNA copiado (cDNA) de una cadena complementaria al mRNA molde usado. Posteriormente la misma transcriptasa o la DNA polimerasa I de E. coli convierte el cDNA de simple cadena en un DNA dúplex. El producto final es un DNA de doble cadena que contiene el «gancho» del extremo 5’. Este se degrada por el tratamiento in vitro con la enzima nucleasa S1 (que hidroliza específicamente DNA de cadena única), así se restablece la estructura de doble hélice con los extremos 5´ libres.
¿Cómo encontrar un gen en una biblioteca de DNA recombinante?
Una de las metodologías más usadas para la selección de clones recombinantes, es el empleo de sondas o rastreadores que tengan alguna homología de bases con el gene a seleccionar. En este punto se ve la importancia de los clones de cDNA, pues si previamente se ha caracterizado el producto de transcripción del gene en cuestión, es posible sintetizar y clonar su cDNA. Así, empleando la metodología de hibridación de secuencias homólogos, se puede «pescar» de la geneteca el clon recombinante que porte el gene.

Figura 6. Construcción de un DNA quimérico. Esquema del flujo de procesos moleculares utilizados para obtener mediante la técnica de DNA recombinante clones quiméricos a partir de un DNA foráneo. En los primeros pasos se somete a digestión con la misma Endonucleasa de restricción tanto el DNA foráneo como el plásmido vector. Una vez linearizado el plásmido vector, se mezclan las dos poblaciones de moléculas digeridas que tienen extremos cohesivos complementarios y se incuban con la DNA ligasa del bacteriófago T4. La reacción de ligación produce plásmidos recombinantes conteniendo porciones de genoma foráneo utilizado. A partir de estas bacterias recombinantes que conforman una biblioteca de DNA recombinante, se seleccionan clones recombinantes que son propagados en medios de cultivo apropiados. Posteriormente mediante utilización de una sonda de DNA específica, se busca el clon recombinante que contenga el fragmento homólogo a la sonda. De esta manera se purifican y estudian los plásmidos quiméricos.
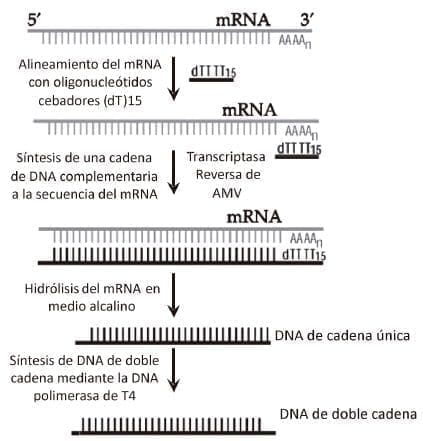
Figura 7. Síntesis de DNA complementario (cDNA) a partir de moléculas de RNA mensajero (mRNA) mediante la utilización de la Transcriptasa Reversa del Virus Mieloblastosis Aviar (AMV). Para que la Transcriptasa inicie la síntesis de una cadena de DNA, es necesario la adición de oligonucleótidos cebadores repetidos (dT)15. Estos forman porciones homologas con las colas 3´de poliadenosinas que tienen los mRNA eucarióticos; de esta manera la transcriptasa reversa completa la síntesis de una molécula de cDNA complementaria. Posteriormente la DNA polimerasa de T4 completa la síntesis de la segunda cadena para producir un sustrato de DNA de doble cadena a ser clonado en vectores apropiados.
En términos prácticos la selección de clones recombinantes se realiza por técnicas de plaqueo de la colección de recombinantes. En resumen esta metodología consiste en: 1. Crecimiento una densidad de colonias apropiadas para individualizarlas en la caja de Petri. 2. Transferencia del DNA de cada colonia a filtros de nitrato de celulosa o de Nylon previa desnaturalización en condiciones alcalinas y posterior neutralización. El DNA de cada colonia es sometido a hibridización con la correspondiente sonda previamente marcada, bien sea radiactivamente o por procedimientos no radioactivos, en condiciones fisicoquímicas apropiadas para que ocurra el apareamiento de porciones de nucleótidos de la sonda con el DNA clonado en el recombinante. Después de una serie de lavados, en condiciones de sal necesarias para retirar los híbridos inespecíficos, (este efecto se denomina astringencia), los filtros son sometidos a procedimientos de revelado con el fin de localizar las colonias que contienen DNA complementario a la sonda empleada (figura 8).
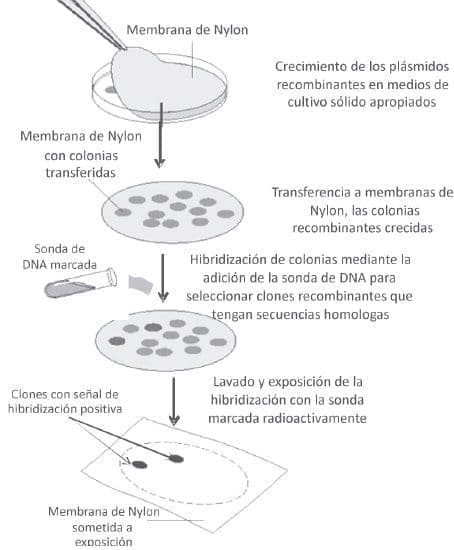
Figura 8. Descripción del método de hibridización en colonias empleado para seleccionar clones recombinantes a partir de una colección inicial de recombinantes. El objetivo es identificar clones recombinantes que tengan fragmentos de DNA complementarios a sondas de DNA específicas más utilizadas. El revelado en la membrana de Nylon, permite volver a la placa con las bacterias y obtener el clon recombinante bacteriano.
Transferencia e hibridización de DNA en «Southern»
Una de las técnicas más utilizadas para identificar y estudiar estructuralmente genes que han sido clonados, es la técnica de transferencia e hibridización en Southern desarrollada por Southern en 1968. Esta metodología combina la separación electroforética de moléculas de DNA en geles de agarosa con la posterior transferencia de éstas a membranas de Nitrocelulosa (NC) o de Nylon.
Es importante destacar que solamente el DNA de cadena única se une a la membrana de manera estable, por lo cual hay que tratar los fragmentos con soluciones desnaturalizantes que permitan la generación de fragmentos de cadena única. Al transferir los fragmentos, separados de acuerdo con su tamaño molecular, a membranas NC es posible hibridizarlos utilizando para ello una sonda de DNA que tenga secuencias complementarias al gene que se desea estudiar. Las condiciones son muy semejantes a las previamente descritas para la hibridización de colonias (figura 9).
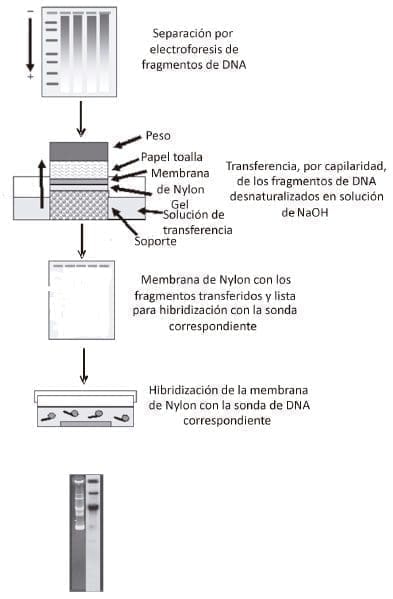
Figura 9. Representación esquemática del flujo de procesos que se desarrollan en el método de transferencia de DNA e hibridización con sonda específica. La metodología se denomina hibridización en Southern.
Recientemente se han desarrollado varias metodologías de transferencia de otras moléculas que tienen casi el mismo principio del Southern. El Northern es la técnica de transferencia de poblaciones de RNA a membranas de NC o de Nylon. El Western es la forma de transferir proteínas de geles de poliacrilamida a membranas de NC, proteínas de geles de poliacrilamida a membranas de NC. A medida que se van perfeccionando las técnicas de detección, se hace más fácil aislar y estudiar genes. En los últimos años ha sido perfeccionada una de las metodologías más potentes, la síntesis de secuencias de oligo-nucleótidos in vitro. Empleando este enfoque, es posible sintetizar cualquier sonda, pues basta conocer parcialmente la secuencia de aminoácidos de una determinada proteína. Esta sonda sirve de base para aislar el gene de una geneteca.
La secuenciación del DNA
La secuenciación de DNA incluye un conjunto de métodos y técnicas bioquímicas cuya finalidad es la determinación del orden de los nucleótidos (A, C, G y T) en un oligonucleótido de DNA. Determinar la secuencia de DNA es útil en el estudio de la investigación básica de los procesos biológicos fundamentales, así como en campos aplicados, como la investigación forense. El desarrollo de la secuenciación del DNA ha acelerado significativamente la investigación y los descubrimientos en biología.
Las técnicas de secuenciación actuales permiten realizar esta secuenciación a gran velocidad, lo cual ha sido de gran importancia para proyectos de secuenciación a gran escala como el Proyecto Genoma Humano. Otros proyectos relacionados, en ocasiones, fruto de la colaboración de científicos a escala mundial, han establecido la secuencia completa de DNA de muchos genomas de animales, plantas y microorganismos.
Aplicaciones de la técnica de DNA recombinante
Aunque las aplicaciones de la Ingeniería Genética cubren casi la totalidad de los procesos en plantas y animales, la medicina y la salud humana, son quizá los campos de aplicación más beneficiados con el descubrimiento y la implementación de la tecnología de DNA recombinante, puesto que esta ha permitido descubrir y buscar alternativas y tratamientos para numerosas enfermedades, la detección temprana de enfermedades genéticas, la producción de sustancias y medicamentos revolucionarios y como complemento a todo esto se ha desarrollado también terapias génicas para algunas de las enfermedades que aquejan diariamente a miles de personas.
Es importante resaltar que aunque existe una gama muy importante de productos de DNA recombinante que tienen usos benéficos para la especie humana y el planeta, se vislumbra una posibilidad real de que algunas de las aplicaciones generen productos nocivos o destructivos para el hombre y el planeta. Nos referimos a una realidad que existe en el armamento biológico almacenado en algunas de los laboratorios de investigación y aplicación en muchos de los países desarrollados y que son más peligrosos que las mismas armas nucleares de destrucción masiva. Por esta razón y desde 1976, año en que se llevó a cabo la Conferencia de Asilomar en California, existen lineamientos bioéticos y de seguridad biológica obligatorios para desarrollar experimentos de DNA recombinante.
