El concepto de gen ha hecho parte fundamental del desarrollo del pensamiento biológico de los dos últimos siglos. Esto porque su definición es fundamental en los campos de la genética, la biología molecular, la evolución y recientemente en la genómica.
La definición de gen fue evolucionando a medida que la biología encontraba nuevas evidencias sobre su estructura y función. Es así que el concepto de gen, lo podemos enmarcar en dos categorías principales: la abstracta y la física. El concepto abstracto se refiere principalmente a ciertos rasgos dentro de los que podemos incluir muchas de las enfermedades conocidas como monogénicas o de herencia simple mendeliana. En este sentido muchos de los biólogos evolucionistas y genetistas humanos clásicos utilizaron este concepto en una unidad de transmisión hereditaria. El concepto abstracto de gen se valida en lo que denominamos fenotipo, o sea el efecto biológico o rasgo detectable.
Con el desarrollo de la química de la herencia, a partir de los años cincuenta del siglo pasado, surge el concepto físico de gen, que lo define como una porción de DNA que está delimitado por una serie de secuencias consenso; en esa secuencia de nucleótidos delimitada, existe un mensaje que se expresa en una macromolécula funcional la cual es una proteína. Así podemos definir el gen desde el punto de vista físico como una secuencia de DNA que se transcribe a un RNA mensajero (mRNA) y del cual se sintetiza un producto funcional. Sin embargo esta definición tan exacta, tiene inconvenientes de ser general, a la luz de los nuevos hallazgos sobre el genoma humano y otros genomas de organismos tanto procarióticos como eucarióticos. Si miramos los nuevos conceptos con relación al papel que juega el RNA en la fisiología celular, la definición anterior queda corta pues al incluir un producto funcional, una proteína, excluye cualquier otro tipo de funcionalidad dentro de la célula.
Si usamos una definición más general, un gen es una secuencia de DNA que es transcrita, existen diferentes tipos de genes: aquellos que codifican por secuencias lineares de proteínas y los genes que transcriben moléculas de RNA diferentes a los mRNA. En estos últimos, existe una gama muy amplia de RNA cuyas funcionesno son solo participativas en la síntesis de proteínas si no en funciones tales como modificaciones postranscripción y replicación del DNA, reguladores, y como RNAs parásito. Todos ellos son transcritos a partir de regiones del genoma (genes RNA) de los organismos o son parte del procesamiento de transcritos de RNA.
El gen eucariótico
A diferencia de la estructura simple de un gen bacteriano, el cual contiene una secuencia promotora desde la que se inicia la transcripción de las secuencias de nucleótidos que codifican por la proteína, la organización de los genes de los organismos eucarióticos es mucho más compleja y flexible (figura 3).
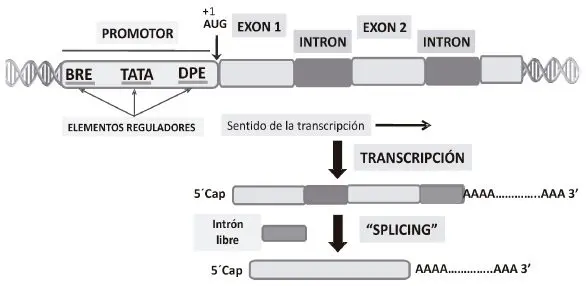
Figura 3. Estructura general de un gen eucariótico. El promotor que es una región del genoma localizada antes del gen propiamente dicho, está compuesto por una serie de secuencias consenso que regulan tanto el inicio de transcripción como los niveles de de síntesis de mRNA en la célula. Este elemento promotor se localiza «upstream» y no se incluye en el transcrito inicial. DPE, TATA y BRE son elementos que controlan la iniciación de la transcripción y están localizados dentro del promotor. En el sentido de transcripción «downstream» se localiza el gen que está divido en exones (secuencias que codifican por aminoácidos) e intrones, las cuales son espaciadoras de los exones. Durante el proceso de transcripción y procesamiento, se sintetiza inicialmente un transcrito primario que contiene tanto intrones como exones. Los intrones son removidos mediante el proceso de «splicing», empalmándose los exones para generar el mRNA que contiene una cola 3’ de poliA y un nucleósido metilado el capuchón en su estremo 5’. Cuando se completan estos tres procesos, el mRNA está listo para su salida del núcleo al citoplasma y su traducción en los ribosomas.
De acuerdo con lo que se muestra en la figura 3, el gen eucariótico posee elementos de control de su propia transcripción; estos se localizan en la región «upstream» denominada promotor. Desde el punto de vista funcional, el promotor contiene una serie de secuencias reguladoras de la transcripción a nivel de tejido específico y también de regulación temporal. Además, más allá, corriente arriba del promotor, existen una serie de secuencias que se denominan potenciadoras (enhancers), que tienen como papel esencial mantener niveles cuantitativamente regulados de transcripción. Es fundamental diferenciar el promotor de bacterias del de los organismos eucarióticos. En estos últimos, se ha producido una proceso de evolución hacia una mayor complejidad pero ganando en funcionalidad y adaptabilidad.
La región codificante del gen eucariótico está conformada por exones y está interrumpida por secuencias no codificantes o intrones. Aunque no es del caso entrar a discutir para qué son los intrones, ellos están en casi todos los genes eucarióticos que codifican por proteínas, con muy pocas excepciones. Podemos entonces afirmar que los intrones han hecho parte de la evolución del gen eucariótico. Durante el proceso de transcripción del gen, se produce un transcrito primario que contiene tanto exones como intrones. Sin embargo, los intrones son removidos y los exones unidos covalentemente, en un proceso denominado «splicing». En este proceso de remoción de intrones y unión de exones, participan una serie de ribonucleoproteinas («SNURP») que poseen un grupo especial de moléculas pequeñas de RNA denominadas URNA. Finalmente el proceso termina con el transporte de una ribonucleoproteína mensajera al citoplasma a través del complejo del poro nuclear.
Los genes pueden ser aislados y clonados mediante Ingeniería Genética
En 1973 A. C Chang, Stanley Cohen y Herber Boyer, hicieron público el primer experimento de DNA recombinante al unir un fragmento producido por la digestión con una enzima de restricción de un plásmido bacteriano de la bacteria Escherichia coli en el sitio de restricción homólogos de un plásmido completo. Un año más tarde, A. C Chang y Stanley Cohen aislaron fragmentos de DNA del cromosoma bacteriano de Staphylococcus y los ligaron covalentemente a un plásmido de E. coli, produciendo plásmidos quiméricos no conjugativos. Cuando estos plásmidos recombinantes se introdujeron en la bacteria E. Coli, lograron que los genes contenidos en los fragmentos de DNA de Staphylococcus se replicaran correctamente en este nuevo sistema genético de E. coli. Los resultados obtenidos en ese entonces, abrieron las puertas para el estudio detallado de la estructura y funcionamiento de genes tanto eucarióticos como procarióticos. Podemos concluir que la tecnología de la manipulación de la molécula de DNA ha avanzado dramáticamente en muy pocos años, llegando a tener una influencia no solo en la ciencia básica sino en las aplicaciones biotecnológicas que han revolucionado la industria y la economía mundiales.
La técnica del DNA recombinante
La metodología que permite reconstruir moléculas de DNA por unión de secuencias denucleótidos de una gran variedad de organismos a un sistema genético de replicación autónoma denominado vector, se describe como Ingeniería Genética o Técnica del DNA Recombinante (el término ingeniería genética es coloquial siendo más apropiado describirlo como DNA recombinante). El desarrollo de la técnica del DNA recombinante fue posible gracias a los estudios previos efectuados durante treinta años en la exploración molecular de los procesos genéticos y celulares. Por esta razón se ha convertido en una serie de metodologías que tienen como objetivo poder manipular de manera parcial la información genética de los organismos.
Sin embargo como cualquier tecnología, la Ingeniería Genética, requiere del empleo de una serie de procesos biológicos controlados y de herramientas moleculares para poder llevar a cabo experimentos en este campo. Por esta razón decimos que la técnica del DNA recombinante necesita para su aplicación de una «caja de herramientas», dentro de las que se incluyen moléculas de DNA vectoras, enzimasque hidrolizan el DNA (Endonucleasas de Restricción), enzimas que polimerizan del DNA (DNA polimerasas), aquellas que ligancovalentemente moléculas de DNA (DNA ligasas) y otras que tienen como objetivo completar los procesos de clonación molecular. A continuación haremos una descripción general de esta caja de herramientas moleculares y cómo se pueden obtener resultados aplicables tanto a la investigación básica como a aquella que involucra una mejora en el funcionamiento de los organismos especialmente en los humanos.
Endonucleasas de restricción los «escalpelos moleculares» para cortar el DNA
La técnica del DNA recombinante utiliza de manera exitosa las endonucleasas de restricción; estas son enzimas bacterianas cuya expresión es inducida por la infección de ciertos bacteriófagos a cepas específicas de bacterias. Este proceso definido como restricción/modificación tiene como resultado la síntesis de una endonucleasa de restricción que «ataca» y fragmenta el DNA infectante. Por otro lado, una enzima de modificación, generalmente una que adiciona grupos metilo a ciertas bases nitrogenadas denominada DNA metilasa, protege el DNA bacteriano de ser destruido por su propia endonucleasa de restricción.
Todas las endonucleasas de restricción reconocen secuencias específicas de nucleótidos sobre un sustrato que es una molécula de DNAde cadena doble. Existen tres clases principalesde estas enzimas denominadas ER I a III. Lasendonucleasas de restricción de las clases I y III son enzimas que reconocen una secuencia específica larga de ocho o más nucleótidos, pero su acción hidrolítica endonucleásica se realiza en algunos nucleótidos externos a este sitio de reconocimiento. Todas las enzimas del tipo I requieren para su actividad nucleásica, de la hidrólisis de una molécula de ATP, además de S-adenosilmetionina (SAM) y Mg++ como cofactores. Debido a que su mecanismo de corte es bastante amplio, este grupo de endonucleasas no son útiles para las experiencias de DNA recombinante. En este sentido, las endonucleasas de restricción de la clase II son, por su especificidad, las empleadas en todos los experimentos de clonación molecular.
Las endonucleasas de clase II, se convierte en una especie de «bisturí molecular« que corta el DNA en sitios específicos los cuales contienen secuencias de nucleótidos que varían de cuatro a seis pares de bases. La secuencia del sitio blanco, determina la especificidad de reconocimiento de la correspondiente endonucleasa de restricción; actualmente se conocen aproximadamente 350 diferentes enzimas de restricción que poseen especificidades de secuencia muy variables.
Para la clasificación y sistematización de las ER, su denominación taxonómica contiene características de la especie bacteriana o cepa de la cual fue aislada. Por ejemplo Eco RI es una enzima de E. coli que contiene el plásmido R. Tal como hemos descrito anteriormente las endonucleasas de restricción del tipo II siempre reconocen secuencias de 4 a 6 nucleótidos; en la tabla 1 se presentan las características más importantes de algunas de las endonucleasas de restricción más comunes.
Aquellas enzimas de restricción que tienen secuencias de reconocimiento de cuatro pares de nucleótidos generan, en la mayoría de las DNAs analizados, fragmentos cortos, mientras que las que reconocen sitios de seis pares de nucleótidos generan fragmentos largos (es necesario aclarar que estadísticamente una secuencia de cuatro nucleótidos está más representada en un genoma que una de seis nucleótidos). Al analizar la información consignada en la tabla 1, se observa que una característica importante de las secuencias de reconocimiento de estas y otras enzimas de restricción, es su simetría. Por ejemplo: En la secuencia
GAA TTC
CTT AAG
Que corresponde al sitio de unión de la EcoRI, existe un eje imaginario de simetría a partir del cual la secuencia se lee lo mismo en ambas cadenas en la dirección 5’→3’. Este arreglo de secuencias es lo que se define como un «palíndrome».
Algunas enzimas de restricción cortan en la mitad del sitio de reconocimiento y producen extremos romos («flush ends»), tal es el caso de Hae III, Alu I, Sma I y Pov II descritas en la tabla presentada y representadas gráficamente en la figura 4. En otras el corte no se produce en el centro del palindrome sino que la hidrólisis de los enlaces fosfodiester ocurre de 3 a 5 nucleótidos más allá; el efecto es la generación de segmentos muy cortos de DNA de una sola cadena, que pueden aparearse entre sí (figura 4). Estos extremos denominados «pegajosos» («sticky ends») son susceptibles de unirse a cualquier otro fragmento de DNA que hubiese sido cortado por la misma enzima o un isoesquisómero de ella, constituyéndose así en un instrumento ideal para la Ingeniería Genética.
