Vp6 y los Péptidos (280-297) de Vp6 (530-559) de Vp4 en la Entrada de Rotavirus
Guerrero Carlos A1*, Gualtero Diego F.1, Guzmán Fanny1, Rodríguez Luz S.1, Acosta L. Orlando1.
Resumen
El mecanismo de entrada de los rotavirus es un proceso de múltiples pasos el cual no está completamente entendido. La proteína de choque térmico HSC70 (heat shock cognate 70) ha sido propuesta como molécula co-receptora para la entrada de rotavirus.
Mediante inmunoprecipitación identificamos la interacción de las proteínas VP6 y VP4 de la cápside del rotavirus RRV con HSC70 en lisados de células MA104 previamente infectadas con rotavirus, sugiriendo la interacción de HSC70 con más de una proteína viral.
Posteriormente se dedujeron las secuencias 531-554 de VP4 y 280-297 de VP6 en base a secuencias reconocidas que unen a HSC70.
En este estudio determinamos que DLPs y los péptidos sintéticos (280-297) de VP6 y (531-554) de VP4, deducidos teóricamente como de unión a HSC70, inhibían la entrada de los rotavirus RRV, YM y WA de manera dosis dependiente en células MA104 y Caco-2.
Igualmente la adición de los péptidos VP4 y VP6 o de DLPs más los péptidos, a concentraciones de inhibición media máxima, inhibió la infección de las tres cepas de manera aditiva. Sueros hiper-inmunes contra los péptidos bloquearon la infección de partículas infecciosas TLPs.
Además mediante un ensayo in vitro se determinó la interacción de DLPs con HSC70. Estos resultados sugieren que VP6 participa durante el proceso de entrada de rotavirus uniéndose a HSC70.
En resumen, este y otros trabajos confirman la función de la proteína HSC70 como molécula receptora durante la entrada de los rotavirus. Probablemente los dominios 280-297 de VP6 y 531-554 de VP4 son regiones utilizadas por los rotavirus para unirse a HSC70, lo cual apoya el modelo de entrada de múltiples pasos involucrando cambios conformacionales en la partícula viral.
Palabras claves: rotavirus, infección, heat shock cognate HSC70, partículas con doble capa (DLPs), péptidos sintéticos.
Introducción
Los rotavirus son el principal agente etiológico de diarreas en niños menores de cinco años a nivel mundial. Estos virus ocasionan cada año 352000 a 592000 muertes (32). In vivo afecta fundamentalmente el intestino delgado e in vitro infectan líneas celulares epiteliales de origen renal como MA104 e intestinales como Caco-2.
Los rotavirus pertenecen a la familia Reoviridae. Son virus de simetría icosahédrica que carecen de membrana lipídica y se caracterizan por tener tres capas concéntricas de proteína que envuelven un genoma de 11 segmentos de RNA de doble cadena (dsRNA).
La cubierta externa está formada por la glicoproteína VP7, y por la hemaglutinina VP4; ésta última se proyecta de la superficie del virus como espigas de 10-12 nm.
La capa intermedia está formada por VP6, siendo ésta la proteína más abundante en el virión, representando aproximadamente el 51% de la masa viral.
La pared de la nucleocápside o ¨core¨ está constituida por la proteína VP2, que engloban al genoma viral (14). Para que el virus sea infeccioso requiere el clivaje de VP4 por tripsina originando las subunidades VP8 y VP5.
Para que se lleve a cabo la infección de la célula por el rotavirus son necesarias interacciones moleculares entre la célula hospedera y el virus.
Este proceso complejo probablemente ocurre por interacciones secuenciales de las proteínas de la superficie de los virus VP4 y VP7 con al menos moléculas de superficie celular diferentes, dispuestas en microdominios lipídicos «rafts» (1,18,20,24,36).
Entre estas moléculas de superficie celular involucradas en la infección por rotavirus está la proteína de choque térmico HSC70 ha sido propuesta como molécula co-receptora en la entrada del rotavirus, al bloquear la entrada del rotavirus preincubando las células con anticuerpos contra HSC70 o proteína recombinante humana HSC70 (19).
Utilizando péptidos sintéticos de proteínas virales se han sugerido dominios de estas proteínas como sitios de reconocimiento para HSC70 e integrinas (11, 38, 39,40). Estudios dirigidos al desarrollo de vacunas han encontrado elevada protección al utilizar a VP6 como inmunógeno mediante diferentes estrategias (5,7,8,9,10,16,27, 31,34). (Ver: Área Clínica: Estudio Bioquímico y Genético de la Enfermedad Troboblástica Gestacional)
Materiales y Métodos
Células y virus. La línea celular de riñón de mono verde africano, MA104, se cultivó en MEM (Eagle´s Minimal Essential Médium), suplementado con 10% (v/v) de suero fetal bovino (SFB). La línea celular de cáncer de colon Caco-2, se cultivó en DMEM suplementado con 20% de SFB.
Coinmunoprecipitación. Para la inmunoprecipitación de HSC70 con proteínas rotavirales se tomaron alícuotas de lisados de células infectadas con rotavirus (1000 UFF), después de 24 a 48 horas postinfección y se precipitaron mediante ultracentrifugación.
El precipitado se resuspendió en MEM y cada alícuota de 100µl contenía 30µg de proteína que se incubó con 10µl de anticuerpo policlonal de conejo anti-HSC70, (generado mediante HSC70 recombinante humana), utilizando como control de la inmunoprecipitación un anticuerpo no relevante, en este caso, un anticuerpo policlonal de conejo (ab-1) dirigido contra la oncoproteína c-neu (oncogene research).
Las alícuotas se incubaron con el respectivo anticuerpo a 4(C toda la noche y se precipitó con proteína A-sepharosa (50 % p/v) manteniendo en agitación suave por 30 minutos.
El precipitado se lavó tres veces con buffer RIPA (50mM tris (pH 7.5), 150 mM NaCl, 1% Nonidet P-40 (NP-40(, 0.5% deoxicolato de sodio, 0.1% dodecilsulfato de sodio (SDS), con 1mM de fenilmetilsulfonilfloruro (PMSF(), luego se hizo un lavado con buffer 50mM HEPES (N-2hidroxietilpiperazina-N´-ácido etanosulfónico) pH: 7.0, 1% NP-40. Posteriormente se hizo un lavado con NaCl 2M, dos con buffer RIPA y dos con agua destilada.
A cada precipitado se le adicionó 25µL de buffer de muestra de Laemmli 1X y se hirvieron durante 3 minutos. Luego, las muestras fueron separadas por SDS-PAGE y se realizó un «Western Blot» húmedo (mini trans-blot cell de Biorad).
Las membranas se bloquearon una hora con leche descremada en PBS solución 5% (p/v). Luego se incubaron con anticuerpo primario hecho en conejo contra rotavirus, o anticuerpo monoclonal de ratón antiVP6, 1026 (31) (donado por el Laboratorio de Genética Humana de la Pontificia Universidad Javeriana) durante toda la noche.
Luego se lavó la membrana y se incubó con anticuerpo secundario conjugado con biotina y se reveló utilizando el sistema ABC (avidina-biotina-peroxidasa y luminol super signal west dura extended de PIERCE).
Purificación de partículas virales. El lisado viral de células MA104 de la cepa Wa con aproximadamente 106 UFF/ml a 107 UFF/ml (unidades formadoras de focos UFF) se ultracentrifugó a 101500 X g por 1 hora 30 minutos a 4ºC. El pellet fue resuspendido en 1 ml de buffer TSM (Tris 0.01 M, pH 7.4, NaCl 0.15 M, MgCl2 0.001M, CaCl2 1 M).
Luego se adicionó Freón (1,1,2 Triclortrifluoretano) para disociar membranas (1:1/3 vol:vol virus:Freón) y se dio vortex por 5 minutos. Se recolectó el sobrenadante y se repitió el procedimiento tres veces, adicionando nuevo buffer cada vez.
El sobrenadante total recolectado fue ultra centrifugado a 101 500 X g por 1 hora 30 minutos a 4ºC para sedimentar el virus. El sedimento fue resuspendido en TSM y ultra centrifugado a 217 100 X g (rotor Sorvall TST 60.4) por 1 hora a 4ºC en un gradiente de cloruro de cesio (CsCl): 0.5 ml con densidad 1.4157, 1 ml con densidad 1.3039, 0.5 ml con densidad 1.2070 y 0.5 ml con sacarosa 30%.
La banda de TLPs y DLPs fue resuspendida cada una en buffer TSM. La fracción correspondiente a las DLPs, se centrifugó a 101 500 X g por una hora a 4ºC. El pellet fue resuspendido en buffer TSM (sin CaCl2 ni MgCl2) con EDTA 50 mM y se dejó durante 1 hora a 37ºC. Nuevamente se centrifugó a 101 500 X g por una hora a 4ºC para retirar el EDTA y se resuspendió en TSM (sin MgCl2 y CaCl2).
Las partículas virales TLPs y DLPs fueron cuantificadas por espectrofotometría a 260 nm teniendo en cuenta el coeficiente de extinción para este tipo de núcleo partículas y se determinó la concentración de partículas virales teniendo en cuenta la siguiente fórmula:
O.D = K x C (O.D = Densidad óptica, C = concentración mg/ml, K = coeficiente de extinción: 8 para DLP y 6 para TLP).
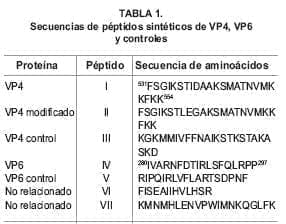
Péptidos. Secuencias peptídicas de VP4 (531-554) y VP6 (280-297) fueron sintetizadas químicamente por la metodología en fase sólida, purificadas por HPLC, caracterizadas por espectrometría de masas y luego liofilizadas.
Como control se sintetizaron péptidos no relacionados y péptidos con el mismo tipo y número de aminoácidos pero con secuencia aleatoria (Tabla 1). Los péptidos fueron resuspendidos en medio MEM, pH 7 y se utilizaron en los ensayos de infectividad.
Ensayos de toxicidad celular. A cada uno de los péptidos se les hizo prueba de toxicidad en células MA104, mediante la técnica de azul de Tripan y MTT como ha sido previamente reportado (30).
Ensayo de infectividad. Inicialmente se hicieron curvas de infección, para hallar la concentración máxima y mínima probable de los péptidos y DLPs con actividad de inhibición de la infección, para esto se tuvo en cuenta trabajos con metodología similar ya reportada (107,106, 19).
Las placas de 96 pozos confluentes con células MA104 o Caco-2 fueron tratadas con diferentes concentraciones del péptido VP4 (0.125 – 4 mg/ml), VP6 (0.015- 2 mg/ml) o DLPs (0.01- 4µg/ ml) por 1 h a 37 °C y 5% de CO2.
En cada placa el tratamiento se hizo por duplicado, en pozos a los cuales se les añadió un volumen total de 30µl. Luego se adicionó una preparación viral que tenía un título de unidades formadoras de foco (UFF) de 106/ ml a 107/ml de un lisado viral, previamente activado con tripsina, de las diferentes cepas de rotavirus (Wa, RRV y YM) para cada uno de los tratamiento y se incubó 1 hora a 37º C en MA104 y Caco-2.
Las placas fueron lavadas dos veces con MEM e incubadas 12 horas a 37°C y 5% de CO2. Posteriormente, las células fueron fijadas con metanol puro, frió, por 45 minutos a 4°C. Las células fueron lavadas tres veces con PBS e incubadas con un anticuerpo policlonal contra rotavirus generado en conejo a partir de virus purificado por CsCl.
Posteriormente, el Ac primario se incubó con un conjugado – HRP (SC 2004, Santa Cruz, Dilución 1:3000 en PBS) por 1 hora a 37°C. En cada paso las células se lavaron dos veces con PBS. Las placas se revelaron con AEC (3-amino-9-ethyl carbazole, SIGMA) y se contaron las UFF en un microscopio invertido (Euromex, Holanda).
Se utilizaron tres controles: el péptido de VP6 o de VP4 con la secuencia aleatoria, un péptido no relacionado y MEM. En todos los experimentos cada virus utilizado se incubó con los diferentes péptidos control.
El porcentaje de infección en todos los casos se relacionó tomando como el 100% los focos contados en los pozos que solamente se realizo la infección en medio MEM.
Producción de Anticuerpos policlonales contra los péptidos sintéticos de VP4 y VP6. Conejos Nueva Zelanda de 3 meses de edad fueron sangrados para obtener suero preinmune. Estos mismos conejos posteriormente fueron inoculados subcutáneamente con 0.5 mg del péptido de VP4 o de VP6 más 0.5 mg del péptido FIS (FISEAIIHVLHSR) junto con adyuvante completo de Freund en la primera inoculación. La segunda y tercera inoculación se aplicó con adyuvante incompleto de Freund cada 20 días.
Finalizado el esquema de inoculación el conejo se llevó a blanco. La sangre se dejó 30 minutos a 37°C y 12 horas a 4°C, posteriormente se centrífugo a 10.000 X g (Beckman) a 4°C durante 30 minutos para recuperar el suero que se mezcló con glicerol (1:1 vol:vol) y azida al 0.02% y se almacenó a -20 °C hasta su uso. Para cada péptido se inocularon dos conejos, generando dos anticuerpos.
Bloqueo de la infección con Anticuerpos contra los péptidos sintéticos. TLPs de la cepa Wa con aproximadamente 106 UFF/ml a 107 UFF/ml, purificadas en gradiente de CsCl, activados previamente, fueron adheridos a células MA104 confluentes a 4ºC durante 45 minutos.
La placa de 96 pozos fue lavada dos veces con MEM e incubada con diferentes diluciones de suero hiper-inmune contra los péptidos VP4 y VP6 a 4ºC durante 30 minutos, luego las células fueron incubadas a 37ºC durante 45 minutos, en una atmósfera de 5% de CO2. Las células se lavaron 2 veces con MEM y se incubaron 12 horas a 37ºC, a una atmósfera de 5% de CO2.
Las células se fijaron y revelaron como se describió anteriormente para el ensayo de infección. Como control se utilizó suero pre-inmune de los mismos conejos y los resultados fueron graficados como porcentaje de infección con respecto al titulo del anticuerpo.
ELISA. Como anticuerpo de captura se usó suero policlonal contra rotavirus hecho en conejo (1:500) y el de detección del antígeno un anticuerpo policlonal contra HSC70 hecho en cabra (0.4 (g/ml, Santa Cruz Biotechnology INC).
Se mezclaron diferentes concentraciones de HSC70 recombinante (0.074 – 1.4 mg/ml) (obtenida del laboratorio del Dr Carlos F Arias del Instituto de Biotecnología de Cuernavaca, UNAM, México) con DLPs (1µg/ml), se incubaron toda la noche a 4 (C en agitación y la mezcla se adicionó sobre el anticuerpo de captura en la placa de ELISA (Nunc) previamente bloqueada con leche descremada al 3% y ovoalbúmina al 2%.
El antígeno con el anticuerpo de captura se incubó toda la noche. Luego se hicieron tres lavados con buffer PBS-Tween 20 (0.05%).
Se adicionó el anticuerpo contra HSC70 en solución de bloqueo PBS-T y se incubó durante una hora a 37ºC. Luego de lavar tres veces la placa se incubó con anti-cabra HRP (1:5000) durante una hora a 37ºC. La placa se reveló utilizando como sustrato OPD (O- Fenilenediamina dihidrocloruro, PIERCE) diluido en buffer para peroxidasa. Las placas fueron leídas como absorbancia a 493 nm en un lector de ELISA. Como control negativo se utilizaron pozos sin HSC70 recombinante.
Análisis de datos. Todos los datos fueron expresados como media y desviación estándar para tres experimentos independientes en los cuales los grupos fueron probados por duplicado en cada uno de los ensayos.
Resultados
HSC70 se asocia con VP4 y VP6. Mediante la técnica de imunoprecipitación se encontró que la proteína HSC70 coinmunoprecipita la proteína viral VP6 identificada mediante un anticuerpo monoclonal (1026). VP4 y VP6 presentan secuencias de unión a HSC70.
Dado que HSC70 interacciona durante la infección con VP4 y VP6 nos propusimos determinar si existían dominios probables de unión de las proteínas virales a HSC70. Para esto, primero se identificaron 40 péptidos reportados en la literatura (2,37) como de unión a HSC70 y su secuencia se alineó con la secuencia de las proteínas virales VP6 y VP4. La alineación de diferentes rotavirus, humanos y de animales, se hizo mediante el programa Clustal X www.evolution.genetics/philyp/.
Realizada la alineación, se seleccionaron secuencias que presentaron características:
1. Alta homología con las proteínas virales.
2. Sobrelapamiento en la misma secuencia viral con más de uno de los 40 péptidos.
3. Conservación de región elegida en los diferentes tipos de rotavirus
4. Exposición de la secuencia en el virión, con respecto a las demás proteínas. Con estos criterios se seleccionó la región (531-554) FSGIKSTIDAAKSMA TNVMKKFKK de VP4, y la región (280-297) IVARNFD TIRLSFQLRPP de VP6 (Tabla 1). Debido a que la secuencia del péptido de VP4 contenía el tripéptido IDA (Ile-Asp-Ala), reportado como de unión a integrina α4β1, la cual ha sido reportada como receptor para la cepa SA11 de rotavirus (22), se sintetizó el péptido II (531-554) de VP4 al cual se le cambio el tripéptido IDA por LEG (Leu-Glu-Gli) que son aminoácidos que pertenecen al mismo grupo funcional. Como péptidos control se sintetizaron péptidos con el mismo tipo y número de aminoácidos, pero con secuencia aleatoria.
Igualmente se sintetizaron péptidos control de secuencia no relacionada. El ensayo de viabilidad con azul de Tripan y MTT mostró que el péptido VP4 (I) tenía un efecto citotóxico en las dos concentraciones analizadas. El péptido II de VP4, IV de VP6 y los de control no tuvieron efecto tóxico.
Los péptidos sintéticos de VP4 y de VP6 inhiben la infección de rotavirus en células MA104 y Caco-2. Para determinar si las secuencias de probable unión a HSC70 presentes en los péptidos sintéticos de VP4 (531-554) y de VP6 (280-297) inhibían la infección de los rotavirus de las cepas Wa, RRV y YM en células MA104 y Caco-2, las células confluentes fueron incubadas con diferentes concentraciones del péptido II (VP4), el péptido IV (VP6) o los péptidos control y posteriormente infectadas con los rotavirus.
En células MA104 y Caco-2 el péptido II de VP4 inhibió la infección de WA, RRV y YM de manera dosis dependiente. En células MA104 la inhibición fue alrededor del 79 % (Wa, RRV) y 68 % (YM) (Fig 1A).
En Caco-2 la inhibición fue del 80% para Wa, 66% para RRV y 68% en YM (Fig 1B). En las células MA104 el péptido IV de VP6 inhibió la infección de las tres cepas de manera dosis dependiente. En MA104 la inhibición fue alrededor del 94 % para RRV y YM y del 74 % en Wa (Fig 1C).
En Caco-2 del 91 % para Wa, 83 % para RRV y 90% en YM (Fig 1D). En las dos líneas celulares los péptidos control no tuvieron ningún efecto en la infección con las tres cepas de rotavirus y en todos los experimentos cada virus utilizado se incubó con los diferentes péptidos control.
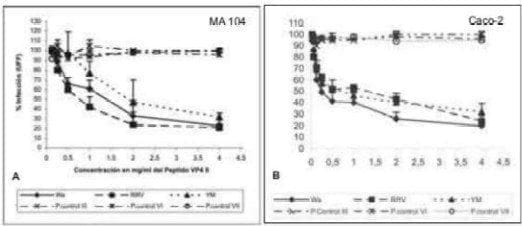
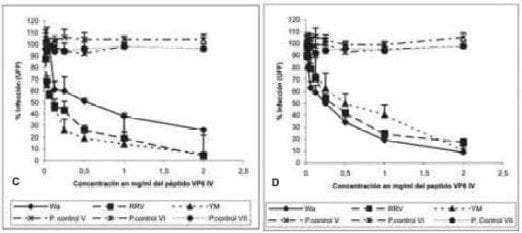
FIGURA 1. Efecto del péptido VP4 y VP6 sobre la infectividad de Wa, RRV y YM. Las células confluentes en placas de 96 pozos fueron incubadas durante una hora a 37 º C con diferentes concentraciones de los péptidos. A y B, Péptido II de VP4 y los péptidos control II, VI y VII con concentraciones desde 0.125 hasta 4 mg/ml. C y D, péptido IV de VP6 y los péptidos control V, VI y VII con concentraciones desde 0.015 hasta 2 mg/ml. Posteriormente las células fueron infectadas con 1000 UFF del correspondiente virus por 1 hora a 37ºC. El exceso de virus fue removido y la infección continuó 14 horas a 37ºC. Las células fueron fijadas y teñidas por inmunocitoquímica. Los resultados son expresados como % de infectividad con respecto a células tratadas con MEM. La curva representa los resultados de al menos tres experimentos independientes cada uno por duplicado.
Como control experimental, se determinó que los péptidos sintéticos no tienen efecto en el ciclo de replicación viral, dado que al infectar primero las células con el virus y luego incubarlas con las diferentes concentraciones de los péptidos no hubo diferencias, respecto a las células incubadas solamente con MEM (datos no mostrados). Estos resultados permiten sugerir que los péptidos compiten con los viriones por moléculas receptoras inhibiendo de esta manera la entrada del rotavirus.
Las DLPs inhiben la infección de rotavirus en células MA104 y Caco-2. Puesto que el péptido sintético de VP6 (280-297) inhibe la infección de los rotavirus quisimos determinar si VP6, que conforma las partículas de doble capa (DLPs), no infecciosas, era capaz de inhibir la infección durante el proceso de entrada del rotavirus.
Para esto, células confluentes MA104 y Caco-2 fueron incubadas con diferentes concentraciones de DLPs de RRV, purificadas por gradiente de CsCl y posteriormente se desafiaron con cada uno de los rotavirus (Fig. 2). En células MA104 las DLPs inhibieron la infección de Wa, RRV y YM de manera dosis dependiente, alcanzando una inhibición alrededor del 81 % para las cepas RRV y YM y de 61% para Wa, cuando todos los ensayos fueron conducidos a una concentración de 3.8 (g/ml de DLPs (Fig. 2A).
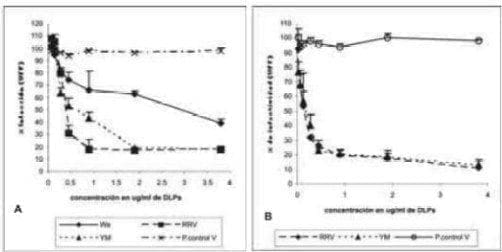
Igualmente, en células Caco-2 las DLPs inhibieron la infección de Wa, RRV y YM de manera dosis dependiente, alcanzando una inhibición de alrededor del 87 % para las cepas RRV y YM cuando los ensayos fueron realizados a una concentración de 3.8 (g/ml de DLPs (Fig. 2B).
Para la cepa Wa se alcanzó una inhibición de 83 % a una concentración de 120 ng/ml de DLPs (Fig. 2C). De esta manera, Wa requiere 30 veces menos concentración de DLPs para alcanzar el mismo porcentaje de inhibición respecto a RRV y YM en células Caco-2, mientras que en células MA104 no se observó esta diferencia en la sensibilidad a la inhibición. El resultado fue similar si las células se lavaban o no luego de incubarlas con DLPs y desafiarlas con los rotavirus.

FIGURA 2. Efecto de las DLPs en la infectividad de Wa, RRV y YM en células MA104 (A) y Caco-2. (B y C) células confluentes en placas de 96 pozos fueron incubadas a 37º C por 45 minutos con diferentes concentraciones de DLPs entre 0.01 hasta 3.8 mg/ml o péptido control V (0.5 mg/ml). Posteriormente se infectaron con 1000 UFF del correspondiente virus por 1 hora a 37ºC. El exceso de virus fue removido y la infección continuo 14 horas a 37º C. Finalmente las células fueron fijadas y teñidas por inmunocitoquimica como se describe en materiales y métodos. Los resultados son expresados como % de infectividad con respecto a células tratadas con MEM. La curva representa los resultados de al menos tres experimentos independientes por duplicado.
Dado que los resultados de inhibición de la infección producidos por la incubación previa de las células con DLPs no excluyen que la inhibición sea una consecuencia de un impedimento estérico asociado a la unión inespecífica y masiva de las DLPs, se realizó un ensayo de infección en células MA104, en el cual se incubaron las células primero con DLPs en cantidades decrecientes y luego se llevó a cabo un desafío con TLPs en una cantidad constante y correspondiente a la utilizada para obtener un grado de infección de referencia asumido como el 0% de inhibición. De esta manera, se obtuvieron relaciones molares entre las TLPs y las DLPs.
Para hallar estas relaciones molares, primero se calculó la concentración de TLPs que generaba entre 4-5 X 106 UFF/ml. Las partículas virales TLPs y DLPs fueron cuantificadas por espectrofotometría a 260 nm teniendo en cuenta un coeficiente de extinción propio para nucleoproteínas con un contenido definido de RNA y se determinó mediante cálculo teórico que para la preparación utilizada los 4-5 X 106 UFF/ml correspondían a 2.1×109 partículas/ml de TLPs.
Igualmente se realizaron los cálculos para determinar el número de partículas de DLPs. De esta manerase realizó un ensayo de infección teniendo en cuenta la relación molar TLPs/DLPs (Fig 3). El resultado mostró que las DLPs bloquearon la infección de las TLPs en un 98%, 62 % y 13% a una relación molar TLP/ DLP de 0.25, 8 y 16, respectivamente.

FIGURA 3. Inhibición de la infección de TLPs a diferentes soluciones molares de TLP/DLP. Células MA104 en cajas de 96 pozos fueron incubadas con DLPs desde 0.78mg/ml (relación 0.25) posteriormente fueron infectadas con 0.32 mg/ml de TLPs previamente activadas con tripsina. Los datos son presentados como % de infección. Como control se utilizo TLPs tratados con anticuerpo neutralizante (27, 36) y células tratadas con MEM. La relación molar se obtiene de dividir el número de partículas teóricamente calculadas de TLPs sobre las DLPs (tabla).
Estos resultados apoyan que el efecto inhibidor en la infección de las cepas Wa, RRV y YM con las DLPs no es debido a un impedimento estérico por moléculas receptoras que utilizan las DLPs, y que de alguna manera interfieren muy probablemente con la unión de las TLPs.
La suma de péptidos sintéticos o de péptido y DLPs inhibe la infección de rotavirus en células MA104 y Caco-2. Quisimos determinar si los péptidos se están uniendo a una misma molécula receptora o a diferentes receptores.
Para esto, utilizamos la concentración media inhibitoria de cada péptido para que teóricamente permaneciera libre la mitad de las moléculas receptoras de la superficie de la célula. Si se unen a una misma molécula receptora la adición de los dos péptidos, o un péptido y DLPs, saturaría nuevamente todos los sitios de unión posibles del mismo receptor restaurando el valor máximo. Si se une a diferentes receptores no debería presentarse efecto aditivo.
En este experimento se utilizó la concentración de inhibición media-máxima de cada péptido y de las DLPs. Células MA104 confluentes fueron incubadas con 1mg/ ml del péptido II más 0.25 mg/ml del péptido IV y posteriormente infectadas con rotavirus.
La suma de los péptidos II y IV a concentraciones de inhibición media-máxima tuvo un efecto inhibidor aditivo del 75 % para la cepa Wa y del 83 % para la cepa RRV. Para la cepa YM no se encontró efecto inhibidor aditivo al utilizar la suma de los péptidos, presentándose una inhibición de sólo 28% (datos no mostrados).
En células Caco-2, la suma de los péptidos tuvo un efecto inhibidor aditivo del 83 % para las cepa Wa, del 82 % para RRV y del 73 % para la cepa YM (datos no mostrados).
Estos resultados muestran que la suma de péptidos aconcentraciones de inhibición media-máxima para las tres cepas de rotavirus en Caco-2 y en MA104 tiene un efecto aditivo, alcanzando porcentajes de inhibición similares a los obtenidos con las concentraciones máximas de cada uno de los péptidos individuales, excepto para la suma de los péptidos VP6 y VP4 en las células MA104 frente a la infección con la cepa YM.
Las células tratadas con péptido control III más el péptido IV confirman que la concentración de inhibición media máxima de los péptidos II y IV permiten la infección con valores cercanos al 50 % de la inhibición que presentaron las tres cepas de rotavirus utilizadas. Igualmente, células MA104 y Caco-2 fueron incubadas con el péptido II más DLPs o péptido IV más DLPs a concentraciones de inhibición media-máxima y posteriormente infectadas con rotavirus Wa, RRV y YM.
La suma de DLPs más el péptido II bloqueó la infección de las cepas Wa, RRV y YM en porcentajes del 81%, 82% y 72%, respectivamente, en células MA104 y del 82%, 78% y 76% para Wa, RRV y YM, respectivamente, en células Caco-2. Similar a lo observado en el ensayo de suma de peptidos, la cepa Wa fue más sensible al bloqueo en la infección en Caco-2 que las cepas RRV y YM.
La suma de DLP más el péptido IV bloqueó la infección en un 79% para las cepa Wa, y alrededor de 84% para las cepas RRV y YM en células MA104 (datos no mostrados. En células Caco-2 se alcanzó un bloqueo de la infección máxima del 91% para Wa, 81% para RRV y del 79% para YM (datos no mostrados).En los cultivos celulares control donde se trató con DLPs más péptido control, se alcanzó aproximadamente un 50% de la inhibición que presentaron todas las cepas virales y en las dos líneas celulares ensayadas.
Al adicionar solamente el péptido control no se presentó efecto inhibidor. Los resultados en las dos líneas celulares utilizadas muestran que la combinación a concentraciones de inhibición media-máxima de partículas DLPs y de los péptidos VP4 y VP6, tiene un efecto aditivo, el cual alcanza porcentajes de inhibición de la infección similares a los obtenidos con la aplicación individual de estos inhibidores a concentraciones máximas.
Los rotavirus utilizan los dominios 531-554 de VP4 y 280-297 de VP6 durante el proceso de entrada a la célula. Los péptidos sintéticos II y IV que contienen secuencias de VP4 y VP6, respectivamente, tienen un efecto inhibidor sobre la infección por rotavirus, esto sugiere que los rotavirus utilizan tales dominios durante el proceso de entrada a la célula.
Para explorar esta posibilidad se utilizó suero hiperinmune generado contra estos péptidos sintéticos de la región 531-554 de VP4 y 280-297 de VP6 para determinar si los anticuerpos bloqueaban la infección. Células MA104 confluentes fueron incubadas con TLPs de la cepa Wa a 4ºC y luego incubadas con diferentes diluciones de suero hiper-inmune que reconoce los péptidos II de VP4 y IV de VP6. Como control se utilizó TLPs incubadas con suero pre-inmune (Fig 4).

FIGURA 4. Anticuerpos contra los péptidos VP4 y VP6 inhiben la infección de rotavirus. Células MA104 en placas de 96 pozos fueron incubadas a 4 ºC por 30 minutos con TLPs (0.32 mg/ml) previamente activados con tripsina. A la misma temperatura se adicionó suero hiper-inmune contra los péptidos VP4 o VP6 o sus pre-inmunes durante 30 minutos. Luego las células se incubaron a 37 ºC por 1 hora, se lavaron y se incubaron 12 horas a 37 ºC. Las células fueron fijadas y teñidas por inmunocitoquimica como se describe en materiales y métodos. Los resultados son expresados como % de infectividad con respecto a células tratadas con MEM. La curva representa los resultados de al menos tres experimentos independientes cada uno por duplicado. Los resultados fueron reproducibles con los dos anticuerpos generados contra cada uno de los péptidos.
Los sueros hiper-inmunes contra los péptidos VP4 y VP6 bloquearon la infección de las TLPs alrededor del 45% y 57%, respectivamente. Como control experimental, se determinó que los anticuerpos no tienen efecto en las células dado que al infectar primero las células con el virus y luego incubarlas con los anticuerpos (pre e hiper inmunes) no hubo diferencias, respecto a las células incubadas solamente con MEM.
Los sueros pre-inmunes no tuvieron efecto en la infección de las TLPs. Los resultados fueron reproducibles con dos anticuerpos, generados en dos conejos diferentes, contra cada uno de los péptidos. Estos resultados, junto con los de inhibición obtenidos con los péptidos sintéticos de VP4 y VP6, apoyan la hipótesis que los rotavirus utilizan los dominios 531-554 de VP4 y 280-297 de VP6 durante el proceso de entrada a la célula.
Mediante un ELISA indirecto se confirmó que los sueros hiper-inmunes, generados contra cada uno de los péptidos, reconocieron sus respectivos antígenos sintéticos. Igualmente, el anticuerpo generado contra el péptido II reconoció las partículas virales TLPs y el generado contra el péptido IV reconoció las DLPs (datos no mostrados).
Ambos anticuerpos también reconocieron los antígenos presentes en células infectadas, tanto por inmunocitoquímica como por fluorescencia, aunque con una intensidad relativamente baja respecto al policlonal utilizado contra rotavirus en la determinación las UFF (datos no mostrados).
Estos resultados sugieren que los dominios 531-554 en VP4 de las TLPs y 280-287 en VP6 de las DLPs están expuestas en la partícula viral y participan en alguna forma en el proceso de infección.
Las DLPs interactúan con la proteína de choque térmico HSC70. HSC70 se ha detectado en superficie celular en células MA104 y Caco-2. Además se ha determinado su asociación con TLPs mediante ensayos ELISA (19). Los resultados de bloqueo de la infección por rotavirus producido por DLPs sugieren que estas partículas se adhieren a moléculas receptoras, interfiriendo así el proceso de entrada del rotavirus.
En este contexto se quiso determinar la unión de VP6, que conforma las DLPs, con HSC70 recombinante mediante un ELISA en sándwich (datos no mostrados). Como control se midió la interacción TLP-HSC70 en las mismas condiciones, evento que ya ha sido confirmado (19). El resultado muestra una interacción dependiente de la concentración de HSC70 con DLPs y TLPs, siendo mayor la interacción con las partículas TLPs. Estos resultados confirman que VP6 se une de manera específica a HSC70.
Discusión
Los resultados de coinmunoprecipitación de HSC70 conjuntamente con VP6 presente en lisados virales sugieren que HSC70 se une a VP6 en algún momento del proceso infeccioso. Proponemos que al incubar los péptidos sintéticos con las células, estos compiten por receptores de rotavirus, lo cual explicaría el efecto inhibitorio observado durante los ensayos de infección.
No obstante, no sabemos qué moléculas de la superficie celular estarían implicadas en la unión, pero dado que estas secuencias de aminoácidos fueron escogidas por su potencial unión a HSC70, muy probablemente se estén uniendo a la célula a través de este receptor.
Con esta metodología no podemos determinar directamente si estas regiones las utiliza el rotavirus durante el proceso infeccioso para unirse con los receptores celulares.
Sin embargo, al incubar anticuerpos, generados contra estos péptidos sintéticos, con los cultivos donde el virus se encontraba previamente adherido a 4˚C a la célula como ha sido descrito (18), se observó que dichos anticuerpos bloquearon la infección, lo cual sugiere que los rotavirus utilizan la secuencia 531-554 de VP4 y 280-297 de VP6 durante la entrada, mediante la exposición de estos dominios en pasos posteriores a la unión inicial del virión con las primeras moléculas receptoras.
De otro lado, los anticuerpos dirigidos contra estas secuencias reconocieron respectivamente a las DLPs y a las TLPs mediante la técnica de ELISA. Los péptidos sintéticos inhibieron más eficientemente a RRV y YM con respecto a Wa en células MA104, pero en células Caco-2 la cepa Wa fue más sensible a la inhibición.
Esto sugiere que las moléculas receptoras para las cepas virales utilizadas están en diferente número o disposición en las diferentes líneas celulares, sin excluir que las proteínas estructurales del virus implicadas en el reconocimiento y la unión también puedan presentar diferencias propias de cada cepa (8, 9,25, 26 29).
La inhibición de la infección producida por las DLPs se podría explicar probablemente por su unión a HSC70, lo cual tendría soporte en el hecho de que in vitro, mediante la técnica de ELISA, estas partículas se unen a la proteína recombinante HSC70, y porque a su vez esta proteína coinmunoprecipita con VP6, lo cual en conjunto confirma la interacción entre estas dos proteínas; igualmente las DLPs de las tres cepas purificadas inhibieron la infección de manera dosis dependiente.
Los ensayos de relación molar DLPs/ TLPs descarta que la inhibición de la infección por las DLPs sea debida a un efecto estérico y sugieren que la unión de las DLPs a moléculas receptoras de la célula interfiere la unión específica de las TLPs, muy probablemente en un segundo paso durante el evento de unión de la partícula rotaviral a la célula.
Por otra parte, como se ha reportado en otros estudios, en la prueba de ELISA, las TLPs se unen a HSC70 con mayor intensidad que las DLPs (19). Esto puede estar ocurriendo porque las TLPs tienen dos sitios posibles de unión a HSC70, los dominios 642-658 (39) y 531-554 de VP5, los cuales exponen durante el proceso infeccioso, mientras las DLPs aparentemente presentan menos sitios de unión a HSC70.
Los ensayos de infección con la combinación de péptidos o la combinación de un péptido y DLPs mostraron un efecto inhibidor aditivo en células MA104 y Caco-2.
Este resultado sugiere que los péptidos se están uniendo a una misma molécula receptora puesto que al utilizar la concentración media inhibitoria, teóricamente permanece libre la mitad de las moléculas receptoras de la superficie de la célula, de tal manera que la adición de los dos péptidos, o un péptido y DLPs, satura nuevamente todos los sitios de unión posibles del mismo receptor restaurando nuevamente el valor máximo.
Nuestros resultados apoyan la hipótesis de reordenamientos estructurales en la partícula viral durante el proceso de entrada, como acontece con virus que poseen envoltura lipídica, tales como el VIH durante el proceso de unión de gp120 para exponer gp 41 (4,12,15,17,21,28,33) o con virus que carecen de envoltura como reovirus (6) y poliovirus, los cuales se unen a varios receptores para exponer VP1 (3,23,24). Independiente de la forma como entra el virus a la células (penetración directa o endocitosis no caveolar, no clatrina dependiente de rafts), lo expuesto es compatible con cualquiera de los mecanismos propuestos para la internalización de los rotavirus a la célula (13,35).
Bibliografia
1. Arias C. F., Isa P., Guerrero C.A., Méndez. E., Zarate S., López T. and López, S. 2002 Molecular biology of rotavirus cell entry. Arch. Med. Res. 33: 356 – 36.
2. Artigues A., Douglas L. Crawford, Ana Iriarte, and Marino martinez. (1998). Divergent HSC70 binding propierties of mitocondrial and cytosolic aspartate aminotransferase. J.Biol. Chem. 273:33130-33134.
3. Benap, D., Filman, D., Trus, B., Cheng, N., Booy, F., Conway, J., Curry, S., Hiremath,C., Tsang, S., Steven, A., and Hogle, J. Molecular tectonic model of virus structural transitions: the putative cell entry states of poliovirus. J Virol. 74: 1342-1354.
4. Berger, E., Murphy, P., Faber, J. 1999. Chemokine receptors as HIV-1 coreceptors: roles in viral entry, tropism, and disease. Annu Rev Immunol. 17: 657-700.
5. Burns J, Siadat-Pajouh, Krishnaney A, Greenberg H. 1996. Protective effect of rotavirus VP6-specific IgA monoclonal Antibodies that lack neutralizing activity. Science. 272: 104-107.
6. Chandran, K., Farsetta, D., and Nibert, M. 2002. Strategy for nonenveloped virus entry: a hydrophobic conformer of the reovirus membrane disruption. J Virol. 76: 9920-9933.
7. Chen S, Jones D, Fynan L, Farrar G, Clegg J, Greenberg H, and Herrmann J. 1998. Protective immunity induced by oral immunization with a rotavirus DNA vaccine encapsulated in microparticles. J. Virol. 72:5757-5761.
8. Choi A, Basu M, Mcneal M, Clements J, and Ward R. 1999. Antibody-independent protection against rotavirus infection of mice stimulated by intranasal immunization with chimeric VP4 or VP6 protein. J. Virol. 73:7574-7581.
9. Choi A, Basu M, Mcneal M, Flint J, Vancott J, Clements J, and Ward R. 2000. Functional mapping of protective domains and epitopes in the rotavirus VP6 protein. J. Virol. 74:11574-11580
10. Choi, A., Mcneal, M., Basu, M., Bean, J., VanCott, J., Clements, J., Ward, R. 2003. Functional mapping of protective epitopes within the rotavirus VP6 protein in mice belonging to different haplotypes. Vaccine. 21: 761-767.
11. Coulson, B. S., S. H. Londrigan, and D. J. Lee. 1997. Rotavirus contains intergrin ligand sequences and a disintegrinlike domain implicated in virus entry into cells. Proc. Natl. Acad. Sci. USA. 94:5389-5394.
12. Doms, R.,Moore, J. 2000. HIV-1 membrane fusion. Targets of opportunity. J Cell Biol. 151: F9-F14.
13. Dormitzer, P., Nason, E., Prasad, B., Harrison, S. 2004. Structural rearrangements in the membrane penetration protein of a non-enveloped virus. Nature. 430: 1053-1058.
14. Estes, M. K. 2001. Rotaviruses and their replication. Virology . In B. N. Fields and D. M. Knipe and P. M. Howley (ed.), Fields Virology, vol. 2. Lippincott-Raven, Philadephia, USA .1747-1786
15. Eckert, D., Kim, P. 2001. Mechanisms of viral membrane fusion and its inhibition. Annu Rev Biochem. 70: 777-810.
16. Feng N, Lawton J, Gilgert J, Kuklin N, Vo P, Prasad B, Greenberg H. 2002. Inhibition of rotavirus replication by a non-neutralizing, rotavirus VP6-specific IgA mAb. J.Clin. Invest. 109:1203-1213.
17. Gallina, A., Hanley, T., Mandel, R., Trahey, M., Broder, C., Viglianti, G., and Ryser, H. 2002. Inhibitors of protein-disulfide isomerase prevent cleavage of disulfide bonds in receptor-bound glycoprotein 120 and prevent HIV-1 entry. J Biol Chem. 277: 50579-50588.
18. Guerrero, C.A., Selene Zarate., Gabriel Corkidi., Susana López, Carlos Arias. 2000. Biochemical characterization of rotavirus receptor in MA104 cells. J Virol, 74: 93-102.
19. Guerrero, C.A., Bouyssounade, D., Zárate, S., Isa, P., López, T., Espinosa, R., Romero, P., Méndez, E., López, S., Arias, C.F., 2002. Heat shock cognate protein 70 is involved in rotavirus cell entry. J. Virol. 76: 4096- 4102.
20. Guerrero CA, Méndez E, Zarate S, Isa P, López S, Arias CF.2000. Integrin alpha(v)beta(3)mediates rotavirus cell entry. Proc Natl Acad Sci U S A.(26):14644-14649.
21. Haywood, A. N. 1994. Virus receptors: binding, adhesion strengthening, and changes in viral structure. J. Virol. 68:1-5.
22. Hewish, M. J., Y. Takada, and B. Coulson. 2000. Integrins (2(1 and (4(1 can mediate SA11 rotavirus attachment and entry into cells. J. Virol. 74:228-236.
23. Hogle James M. 2002. Poliovirus cell entry: common structural themes in viral cell entrypathways. Annu Rev Microbiol. 677- 702.
24. Isa P, Realpe M, Romero P López S, and Arias C.2004. Rotavirus RRV associates with lipid membrane microdomains during cell entry. Virology. 322: 370-381.
25. Kirkwood, C., Bishop, R., and Coulson, B. 1998. Attachment and growth of human rotavirosis RV-3 and S12/85 in Caco-2 cells depend on VP4. J Virol. 72: 9348-9352.
26. Kitamoto, N., Ramig, D., Matson, D., and Estes, M. 1991. Comparative growth of different rotavirus strains in differentiated cells (MA104, HepG2, and Caco-2). Virology. 184: 729-737. 27. Ludert JE, Ruiz MC, Hidalgo C, Liprandi F. 2002 .Antibodies to rotavirus outer capsid glycoprotein VP7 neutralize infectivity by inhibiting virion decapsidation. J Virol. 76: 6643-6651.
28. Markovic, I., Stantchev, T., Fields, K., Tiffany, J., Tomiç, M., Weiss, C., Broder, C., Strebel, K., and Clouse K. 2004. Thiol/disulfide exchange is a prerequisite for CXCR4-tropic HIV-1 envelope-mediated T-cell fusion during viral entry. Blood. 103: 1586-1594.
29. Mathieu M, Petiptas I, Navaza J, Lepault J, Kohli E, Pothier P, Prasad B, Cohen J, and Rey F. 2001. Atomic structure of the major capsid protein of rotavirus: implications for the architecture of the virion. EMBO J. 20:1485-1497. 30. Mosman T. 1983. Rapid colorimetric assay for cellular growth and survival: aplication of proliferation and cytotoxicity assay. J Immunol Methods. 65:55-63
31. Poncet D, Corthier G, Charpilienne A and Cohen J. 1990. A recombinant vaccinia virus expresisng the major capsid protein of simian rotavirus-induced anti-rotavirus antibodies. Virus Res. 15:267-274.
32. Quan, C. M., and F. W. & Doane. 1983. Ultraestructural evidence for the cellular uptake for rotavirus by endocytosi.Intervirology. 20:223-231.
33. Ryser, H., Levy, E., Mandel, R., and DiSciullo, G. 1994. Inhibition of human immunodeficiency virus infection by agents that interfere with thiol-disulfide interchange upon virus-receptor interaction. Proc. Natl. Acad. Sci. USA. 91: 4559-4563.
34. Schwartz-Cornil I, Benureau Y, Greenberg H, Hendrickson B, and Cohen J. 2002. Heterologous protection induced by the inner capsid proteins of rotavirus requires transcytosis of mucosal immunoglobulins. J Virol. 76:8110-8117. 35. Sanchez-San Martin, C., Lopez, T., Arias, C.F., Lopez, S., 2004. Characterization of rotavirus cell entry. J. Virol. 78: 2310-2318.
36. Shaw, R. D., P. T. Vo, P. A. Offit, B. S. Coulson, and H. B. Greenberg. 1986. Antigenic mapping of the surface proteins of rhesus rotavirus. Virology 155: 434-451.
37. Takenaka I., Leung S., McAndrew S., Brow J., Hightower L. (1995). HSC70-binding peptides selected from a Phage Display peptide Library that Resemble Organellar Targeting Secuences. J. Biol. Chem. 270: 19839-19844
38. Zárate, S., Rafaela Espinosa., Pedro Romero., Carlos A Guerrero., Carlos F. Arias and Susana López. Integrin alpha 2 Beta 1 mediates the cell attachment of the rotavirus neuraminidase-resistant variant nar3. Virology. 2000. 278: 50 – 54
39. Zárate, S., Cuadras, M. A., Espinosa, R., Romero, P., Juárez, K. O., Camacho-Nuez, M., Arias, C. F. y López, S. 2003. Interaction of Rotaviruses with HSC70 during Cell Entry Is Mediated by VP5. J Virol. 77: 7254-7260
40. Zárate, S., R. Espinosa, P. Romero, E. Méndez, C. F. Arias, and S. López. 2000. The VP5 domain of VP4 can mediate the attachment of rotaviruses to cells. J. Virol. 74:593-599.
1 Laboratorio Biología Molecular de Virus, Departamento de Ciencias Fisiológicas, Facultad de Medicina, Universidad Nacional de Colombia. Bogotá, Colombia.
* Dirección de correspondencia del autor:
Departamento de Ciencias Fisiológicas, laboratorio Biología Molecular de Virus, Facultad de Medicina, Universidad Nacional de Colombia.
