Péptidos Sintéticos con cisteínas de secuencias rotavirales inhiben la infección en células MA104
Para realizar una aproximación a la implicación de las proteínas del rotavirus como sustratos del intercambio tiol/disulfu posiblemente catalizado por la PDI, se sintetizaron péptidos sintéticos de los rotavirus RRV y Wa, correspondientes a secuencias de las proteínas VP7 y VP4 que contienen cisteínas.
Las secuencias seleccionadas incluyeron la región constituida por los aminoácidos 200-219 de VP4, las regiones 189-210 y 243-264 de la proteína VP7 de las cepas RRV y Wa.
Como controles se sintetizaron péptidos no relacionados con rotavirus y aquellos rotavirales en los que se sustituyó la cisteína por serina, además de los péptidos con la secuencia de aminoácidos al azar con y sin cisteínas (T 1). (Lea también: Interacción Rotavirus – PDI)
Se evaluó la infección de rotavirus RRV en células MA104 en presencia de diferentes concentraciones de los péptidos mediante inmunocitoquimica midiendo las UFF.
Recientemente se ha reportado que los rotavirus interactúan con la integrina v3 a través de la región 161-169 de la proteína VP7, región peptídica llamada CNP (Péptido 12 o P12 de la Tabla 1); esta secuencia en VP7 es altamente conservada entre las diferentes cepas de rotavirus y bloquea eficientemente la infección de la cepa RRV mas no la unión a la célula (Zarate et al., 2004); este péptido fue utilizado como control positivo de inhibición.
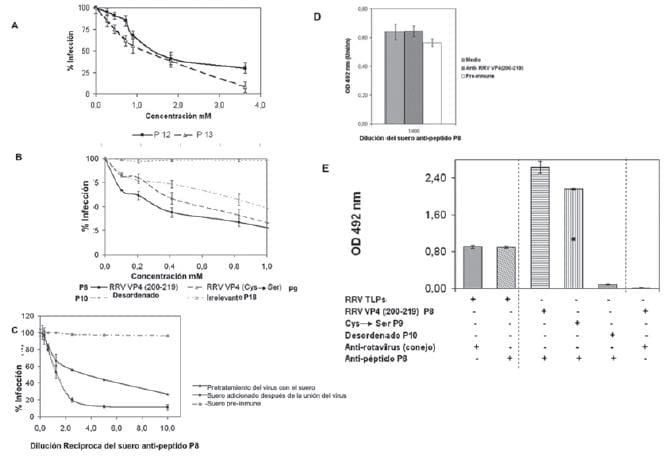
La disminución en el porcentaje de infección para el P12 es dosis dependiente, el 50% de inhibición se logró a una concentración 1,5 mM de péptido. Tanto el péptido original, P12, como su versión modifi cada de Cys por Ser (P13), inhiben la infección por rotavirus con un perfil similar (Fig. 10A), indicando para este caso poca relevancia de ese aminoácido cisteína en el proceso infectivo.
La secuencia de la proteína VP8 (200-219) de la cepa RRV, P8, tiene un efecto inhibitorio de la infección rotaviral dependiente de la concentración, se inhibió el 50% de la infección con una concentración de 0.34 mM de péptido (Fig. 10B).
Para el caso de la versión modificada el P9 aunque tuvo un comportamiento similar de inhibición dependiente de la concentración, el 50% inhibitorio se logró a una concentración de 0.8 mM, esto podría reflejar una mayor afi nidad del P8 por la molécula receptora en la célula.
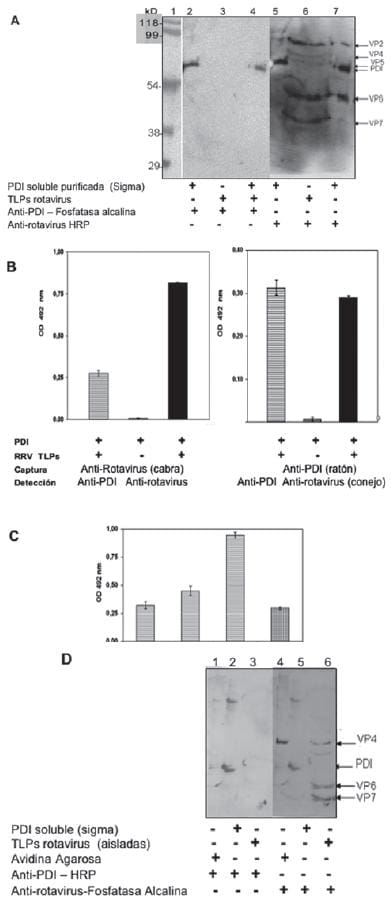
Figura 8 (B y C). Interacción entre PDI y TLPs de RRV. En B, las TLPs se mezclaron con 4 μg de PDI soluble (bovina-Sigma) por 30 min a 20ºC, el complejo se adicionó sobre una placa de ELISA sensibilizada con anti-rotavirus generado en cabra y detectada con anti-rotavirus o anti-PDI generados en conejo.
El delta corresponde a sustracción de la absorbancia del control consistente de células lisadas sin virus, o PDI sin virus. En C, diferentes concentraciones de PDI se adicionaron al virus, en presencia y ausencia de DTNB, siguiendo el procedimiento anterior.
Figura 8D. Interacción entre PDI y TLPs de RRV. Células Caco-2 fueron marcadas con sulfo-NHS-SSBiotin, se adicionó rotavirus Wa por 40 min a 4 °C y 15 min a 20 °C. Las células fueron lisadas y las proteínas biotiniladas inmovilizadas con agarosa-avidina. Las proteínas inmovilizadas se analizaron por “Western blotting” con avidina-HRP y anticuerpos anti-PDI, para detectar PDI y con anti-rotavirus para detectar las proteínas virales.
El lisado celular se adicionó a una placa de ELISA, preincubada con anti-rotavirus (A), o anti-PDI (B) hechos en conejo y se reveló con anti-rotavirus o anti-PDI generados en cabra para detectar los antígenos protéicos y con avidina-HRP para evaluar la incorporación de biotina.
Al parecer estos dos péptidos son de mayor afinidad al ser comparados con el P12, cuya concentración inhibitoria al 50% fue 4.4 veces mayor que la encontrada para P8. Al aumentar la concentración de los péptidos P8 y P9 hasta 1,7 mM se logra un porcentaje de inhibición del 90%; sin embargo el perfil observado a esas concentraciones para los péptidos; sin embargo el perfi l de inhibición de P9 no es saturable correspondiente a aquellos que se describen como de baja especificidad (Fig. 10B).
El efecto de péptidos sintéticos de secuencias no relevantes de rotavirus no produjo inhibición de la infección, sin embargo el P10, un péptido con un contenido de aminoácidos igual al de P8 pero organizados al azar, tuvo un efecto inhibitorio de menor afinidad, alcanzándose el 50% solo hasta 1,2 mM (Fig. 10B).
Anticuerpos generados en conejo contra el P8 fueron capaces de reducir la infectividad por rotavirus en un 90% con un perfil de saturación. Esto se logró cuando los anticuerpos fueron adicionados al complejo células-virus, después del proceso de adherencia del virus a 4ºC.
Cuando el anticuerpo anti-péptido P8 es incubado con el virus y luego esta mezcla adicionada a las células, la inhibición es del 70%, comparados contra el 100% de infección mostrado por el suero preinmune (10C). Este anti-péptido no afecta el proceso de adherencia del virus a la célula según indican los resultados de unión rotavirus-células medido por ELISA de captura (Fig. 10D).
El comportamiento del anticuerpo anti-péptido P8 frente a la infección rotaviral, apoya los resultados encontrados de inhibición de la infección con el péptido P8, también sugieren un efecto neutralizante de este anti-péptido sobre las partículas virales.
El anticuerpo anti-P8 reconoció tanto al P8 como a la versión modifi cada P9, más no a la versión desordenada P10; sin embargo, el péptido P8, al igual que el P9 y el P10 no fueron reconocidos por los anticuerpos anti-rotavirus generados en un conejo inoculado con las TLPs (Fig. 10E).
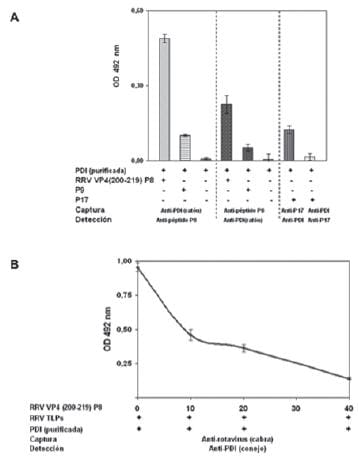
Para determinar la posible interacción entre los péptidos derivados de VP4 y PDI, tanto el péptido como la enzima se mezclaron, la interacción fue analizada en un sistema de ELISA de captura, capturando los antígenos alternativamente con anti-PDI (de ratón) o anti-péptido P8, y detectando la interacción con el anticuerpo reciproco, respectivamente.
Se determinó una fuerte interacción entre el péptido P8 y PDI, la cual disminuyó cuatro veces al cambiar en el péptido las cisteínas por serinas (Fig. 11A), en el que se muestra además el bajo reconocimiento hacia el complejo PDI-P17, por parte de los anticuerpos anti-PDI y anti-péptido P17, siendo este un péptido de secuencia sin cisteínas y no relacionada con rotavirus.
Para confirmar la especificidad de la interacción PDIVP8 mediada por el péptido P8, se realizó un ensayo de competencia medido por ELISA de captura, en el cual se capturaron los antígenos rotavirales y se detectó PDI en interacción con las TLPs de RRV, en presencia de diferentes concentraciones de péptido P8. Los resultados de este ensayo mostraron que a medida que aumenta la concentración de P8 hay menor detección de PDI. Estos resultados sugieren que el rotavirus RRV interacciona con PDI mediante la secuencia de VP8 (200-219) (Fig 11B).
Figura 10. Infección de virus RRV a Células MA104 en presencia y ausencia de péptidos rotavirales, modifi cados y no rotavirales: A. Péptido CNP (P12) de VP7 (161-169) de unión a Integrina v3 y la versión modifi cada P13 (Cys x Ser). B. Péptido de RRV VP4 (200- 219) (o igualmente VP8 200-219); la versión modifi cada P9, la versión de secuencia al azar del original (P10) y la versión al azar del modifi cado (P18). Las monocapas fueron infectadas por rotavirus MOI 1:0,5 en las concentraciones indicadas de péptido.
El porcentaje de infección se midió cuantifi cando el virus como unidades formadoras de foco (UFF) mediante inmunocitoquimica con anti-rotavirus hecho en conejo y el sistema peroxidasa. C. Inmunocitoquimica del tratamiento de las células con el anticuerpo anti-péptido P8 en el proceso infectivo con rotavirus RRV. D. Unión a 4°C de rotavirus RRV a las células en presencia y ausencia del suero del anti-peptido P8.
El virus unido se cuantificó mediante ELISA de captura. E. Reconocimiento especifi co del anticuerpo anti-P8 hacia los peptidos P8, P9 y P10, y hacia las TLPs de RRV; así como también reconocimiento especifico del anti-rotavirus hacia los péptidos. Todo medido mediante ELISA y el sistema peroxidasa-OPD.
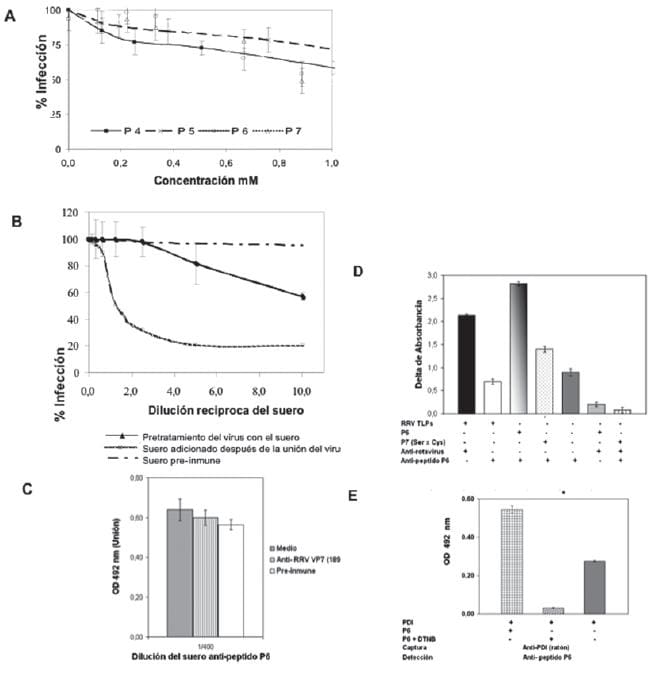
De la proteína VP7 se seleccionaron dos regiones que contienen cisteínas en sus secuencias, y que corresponden a los aminoácidos 189-210 y 243-264. La secuencia peptidica de VP7 189-210 (P6) incluye 3 cisteínas (Cys) y una versión truncada de esta secuencia ubicada ente lo aminoácidos 192 y 210 (P4) y que posee solo 2 cisteínas (Cys). Se estudiaron también las versiones modifi cadas mediante el cambio de cisteína por serina (Cys x Ser), correspondientes repectivamente a los péptidos P7 y P5 (Tabla 1).
Los resultados indican que al adicionar los péptidos a la célula, a una concentración de 2 mM y luego infectarlas, hay inhibiciones de la infección rotaviral hasta en un 70%, con tendencia a un perfil de saturación.
Para alcanzar el 50% de inhibición de la infección se requirió de una concentración de 1.26 mM para el péptido P4 y de 1.09 mM para el péptido P6. Hay un efecto importante de las cisteínas en la interacción con el (los) receptor(es), ya que al analizar las respectivas versiones modificadas (Cys x Ser) el 50% de inhibición de la infección se obtiene prácticamente con el doble de la concentración de péptido original (Fig. 12A)
Anticuerpos generados en conejo contra el péptido P6 fueron capaces de reducir la infectividad del rotavirus en un 90% cuando los anticuerpos fueron adicionados después del proceso de unión (adherencia) del virus a las células a 4ºC.
Cuando los anti-P6 son incubados con el virus y luego esta es mezcla adicionada a las células, el perfil de la curva de inhibición de la infección no es saturable, con un valor de 60% a la máxima concentración de anicuerpos utilizada (Fig. 12B). Los anticuerpos anti-P6 no afectan el proceso de adherencia del virus a la célula a 4 (C, según indican los resultados de unión rotavirus-células obtenidos por ELISA de captura (Fig. 12C).
Los resultados de inhibición de la infección rotaviral con los anticuerpos anti-P6, apoyan los datos obtenidos con los péptidos P4 y P6 con relación a la infección por rotavirus.
Los anticuerpos anti-P6 no presentan un efecto neutralizante sobre las partículas virales antes de su unión a la superficie; mientras que el efecto inhibitorio de estos anticuerpos sólo se expresa cuando son añadidos en un momento posterior a la unión (adherencia) del virus a la célula. Además, no se detectó un apreciable reconocimiento de los anticuerpos anti-P6 hacia las TLPs de la cepa RRV, de acuerdo con las determinaciones realizadas con ELISA (Fig. 12D).
Estos resultados, en conjunto, sugieren que el virus requiere de cambios conformacionales que impliquen la exposición la región 189-210 de VP7 para ser utilizada en las interacciones subsecuentes del virus con las moléculas de superfi cie de la célula.
Los anticuerpos anti-P6 reconocieron los péptidos P4, P6 y en menor medida a los péptidos modificados P5 y P7, mientras que ninguno de estos péptidos fue reconocido por los anticuerpos anti-rotavirus generados en un conejo inoculado con TLPs (Fig. 12D).
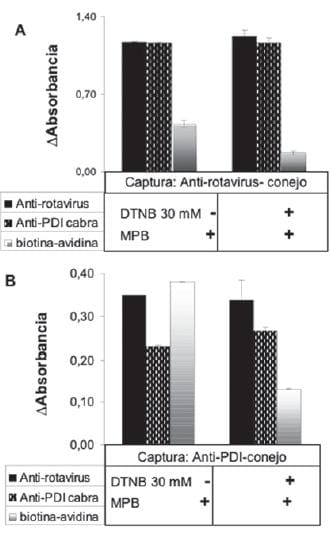
La interacción fue analizada en un sistema de ELISA de captura, capturando los antígenos alternativamente con anticuerpos anti- PDI o anti-péptido P6, y detectando la interacción similares a los obtenidos en ausencia del inhibidor, lo que sugiere que el estatus reductor de la membrana es rápidamente alcanzado siendo el rotavirus sensible a la interacción las proteínas de actividad tiol-disulfuro.
Con estos mismos anticuerpos en forma recíproca.
Figura 11A. Interacción PDI con los Peptidos P8, P9, P17. PDI (4 μg) se mezcló con los péptidos en relación 4 a 1, la interacción fue analizada por ELISA de captura. B. Ensayo de competencia entre el P8 y el rotavirus por PDI. Diferentes cantidades (μg) de PDI se incubaron con el P8 y se adicionó el virus y se detectó PDI, mediante ELISA de captura, con el sistema peroxidasa-OPD.
Los resultados muestran una apreciable interacción de los péptidos que contienen cisteínas con PDI, interacción que es minimizada cuando la reacción se hace en presencia de DTNB o con los péptidos modifi cados por cambio de las cisteínas por serinas (Fig. 12E). Estos resultados sugieren que el rotavirus interacciona con PDI involucrando el sitio catalítico redox de PDI.
Figura 12A. Infección de virus RRV a Células MA104 en presencia y ausencia de los péptidos rotavirales P4 y P6 de VP7 (189-210, 192-210) y la versión modificada P5 y P7 (Cys x Ser). Las monocapas fueron infectadas por rotavirus MOI 1:0,5 en las concentraciones indicadas de peptido.
El porcentaje de infección se midió cuantifi cando el virus como unidades formadoras de foco (UFF) mediante inmunocitoquimica con anti-rotavirus hecho en conejo y el sistema peroxidasa. B. Inmunocitoquimica del tratamiento de las células con el anticuerpo anti-péptido P6 en el proceso infectivo con rotavirus RRV. C.
El virus unido se cuantifi có en presencia del anti-peptido P6 mediante ELISA de captura. D. Reconocimiento especifico del anticuerpo anti-P6 hacia los peptidos P6 y P7, y hacia las TLPs de RRV; así como también reconocimiento especifico del anti-rotavirus hacia los péptidos.
Todo medido mediante ELISA y el sistema peroxidasa-OPD. E. Interacción PDI con los Peptidos P6 y P7. PDI (4 μg) se mezcló con los péptidos, la interacción fue analizada por ELISA de captura.
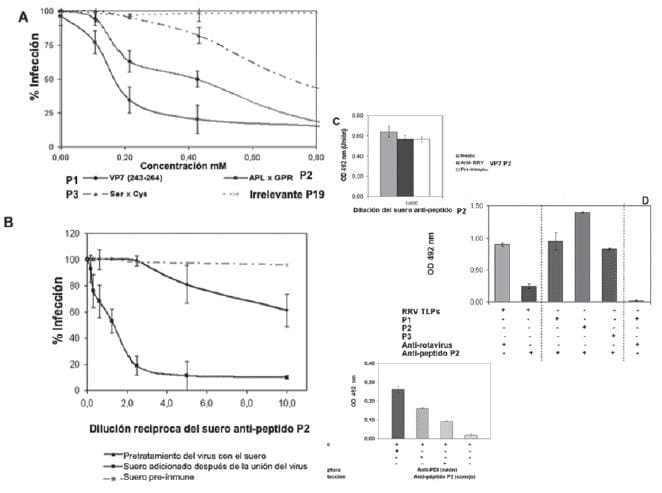
La segunda región de VP7 estudiada corresponde a la secuencia de aminoácidos 243-264, cuyo péptido original (P1) incluye la secuencia de aminoácidos GPR que constituye el ligando de unión a la integrina x2 (Coulson et al., 1997).
Los aminoácidos glicina y arginina fueron reemplazados en el péptido P2 por alanina y leucina respectivamente, se sintetizó también el péptido modificado en el que se cambiaron las cisteínas por serinas, asignado como el péptido P3.
Cuando las células se infectaron con rotavirus en presencia de concentraciones crecientes de estos péptidos, se observó inhibición de la infección de manera dosis dependiente hasta del 85%, con un perfi l de saturación a partir de 0.25 mM para el péptido P2 (Fig. 13A).
Existe una apreciable diferencia de comportamiento frente a la infección mediada por los péptidos P2 y P3, ya que para alcanzar el 50% de inhibición con el péptido P2 se requiere 0.18 mM de péptido, mientras que para su versión modificada Cys por Ser (P3) aumenta hasta 0.82 mM, en una relación de 4,5 veces, reflejando una disminución de la afinidad del péptido por la molécula receptora (Fig. 13 A).
Los anticuerpos generados en conejo contra el péptido P2 redujeron la infectividad por rotavirus en un 90%, cuando se adicionaron al complejo célulasvirus. Esta inhibición ocurrió cuando se adicionó el anticuerpo después que el virus ya estaba adherido a la célula, a 4ºC, en un perfil saturable.
Cuando el anticuerpo anti-P2 fue incubado con el virus y esta mezcla adicionada a las células, la inhibición de la infección fue del 50% a la máxima concentración de suero analizada, en un perfil no saturable que se relaciona con un comportamiento de baja especificidad (Fig. 13B). Al igual que en los casos anteriores, estos anticuerpos contra el péptido P2 no afectan el proceso de adherencia del virus a la célula (Fig. 13C).
Los resultados obtenidos con los anticuerpos anti-péptido P2 en el ensayo de inhibición de la infección, apoyan los resultados obtenidos al incubarestos péptidos con las células, en los cuales P1 y P2 compiten con el rotavirus por los receptores celulares.
Al parecer, de manera similar a lo obtenido para la región de VP7 189-210, la secuencia de VP7 243- 264 no estaría expuesta en la partícula viral. Esto se deduce porque no se observa un efecto neutralizante de los anticuerpos anti-P2 sobre las TLPs, cuando los anticuerpos se incuban con las TLPs a 37 (C y luego se adicionan a la célula.
Estos resultados en conjunto, sugieren que el rotavirus requiere de cambios conformacionales una vez se une a la célula, en lo denominado post-unión, para que esta región 243-264 de VP7 sea expuesta a la superfi cie celular y pueda utilizarse en el proceso de entrada del virus a la célula.
Los anticuerpos anti-péptido P2 reconocieron el péptido P2, y en menor medida a los péptidos P3 y P1, mientras que ninguno de estos péptidos fue reconocido por los anticuerpos anti-rotavirus generados en un conejo inoculado con TLPs (Fig. 13D).
Siguiendo el esquema expuesto anteriormente de incubación de la PDI y los péptidos, analizamos si hay interacción entre PDI y estos péptidos. Los resultados muestran interacción de los péptidos P1 y P2, que contienen cisteínas, con la PDI. La interacción disminuye cuando la reacción se hace incubando PDI con el péptido modifi cado P3 (Cys por Ser) (Fig. 13E).
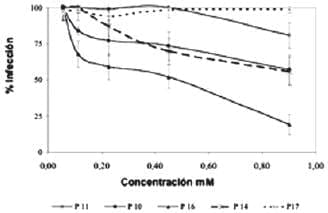
El péptido P11 corresponde a la secuencia de la proteína VP8 (213-232) de la cepa Wa. La secuencia de aminoácidos de esta región carboxiterminal presenta diferencias entre las cepas Wa y RRV. L cepa Wa tiene una serina en la posición 203, mientras que la cepa RRV (y otras cepas dependientes de ácido siálico) contiene cisteína.
En la posición 216 de la VP8 hay una cisteína conservada en las diferentes cepas rotavirales. A1 adicionar el péptido P11, a una concentración 1.57 mM a las células MA104, se inhibió la infección del rotavirus RRV en un 50% (Fig. 14).
Este mismo experimento se llevó a cabo con otros péptidos no rotavirales que tienen cisteínas en su secuencia. En este caso, se presentó inhibición de infección, sugiriendo que la presencia del aminoácido cisteína confi ere capacidad de inhibir la infección por rotavirus. Probablemente hay inhibición porque estos péptidos, a pesar de ser secuencias no relacionadas con rotavirus, también son sustrato de la PDI (Fig. 14).
Los péptidos sin cisteínas y no relacionados con rotavirus, como el P17 y su anticuerpo, no tienen efecto alguno sobre la infección rotaviral y tampoco interaccionan con PDI. Los péptidos P18 y P19, constituidos por secuencias al azar pero compuestas por los aminoácidos de los péptidos modifi cados (Cys x Ser), tampoco tuvieron incidencia sobre la infección rotaviral.
| FIGURA 13 (A y B). En A, Infección de virus RRV a Células MA104 en presencia y ausencia del péptido rotaviral P2 de VP7 (243-264) y las versiones modificadas P2 y P3. Las monocapas fueron infectadas por rotavirus MOI 1:0,5 en las concentracionesindicadas de péptido. El porcentaje de infección se midió cuantifi cando el virus como unidades formadoras de foco (UFF) mediante inmunocitoquimica con anti-rotavirus hecho en conejo y el sistema peroxidasa. B. Inmunocitoquimica del tratamiento de las células con el anticuerpo anti-péptido P2 en el proceso infectivo con rotavirus RRV. | FIGURA 13 (C, D y E). Infección de virus RRV a Células MA104 en presencia y ausencia del péptido rotaviral P2 de VP7 (243-264) y las versiones modificadas P2 y P3. C. El virus unido a la células en presencia de anticuerpo anti-péptido P2, se cuantificó mediante ELISA de captura. D. Reconocimiento específico del anticuerpo anti-P2 hacia los péptidos P1, P2 y P3, y hacia las TLPs de RRV; así como también reconocimiento específi co del anti-rotavirus hacia los péptidos. Todo medido mediante ELISA y el sistema peroxidasa- OPD. E. Interacción PDI con los péptidos P1, P2 y P3. PDI (4 μg) se mezcló con los péptidos en relación 4 a 1, la interacción fue analizada por ELISA de captura. |
Figura 14. Infección de virus RRV a células MA104 en presencia y ausencia de péptidos no relacionados con rotavirus, con y sin cisteinas. Las células MA104 se incubaron con los péptidos por 30 min, luego se retaron con el rotavirus RRV y se incubaron por 12 h. La presencia del virus se evaluó por inmunocitoquimica.
CLIC AQUÍ Y DÉJANOS TU COMENTARIO