Interferencia de la Infección por Rotavirus, Discusión
Las proteínas PDIs se han visto implicadas en importantes procesos biológicos en sitios diferentes al RE, aunque no se conoce mucho acerca de las funciones y los mecanismos de acción de las PDIs, en esos sitios. Al menos tres proteínas de esta familia en mamíferos han sido encontradas en la superficie celular, el espacio extracelular, el citosol y el núcleo (Essex et al 1995; Turano, C. 2002).
A nivel de la membrana celular la PDI ha sido encontrada en diferentes células, entre ellas plaquetas activadas, hepatocitos, páncreas exocrino, células endoteliales, linfocitos CHO, BHK (Chen et al., 1992; Terada et al., 1995; Yoshimori et al., 1990; Hotchkiss et al., 1998). (Lea también: Péptidos Sintéticos con cisteínas de secuencias rotavirales inhiben la infección en células MA104)
En este trabajo reportamos, por primera vez, que PDI se localiza en la membrana citoplasmática de las células MA104 y Caco-2, permisivas a la infección por rotavirus, en una concentración muy inferior a la encontrada al interior de la célula (Fig 4C).
La presencia de PDI en estos sitios puede darse por un mecanismo aún no elucidado de exportación, existiendo rápidamente recambio de ésta; sin embargo, su función en membrana citoplasmática no está aún completamente determinada aunque sigue estando relacionada con las propiedades redox (Schwaller et al., 2003).
La PDI asociada a la superficie celular está implicada en la reducción de los puentes disulfuro de la toxina de la difteria, que por esta vía adquiere la capacidad de entrar en la célula y desplegar su actividad citotóxica (Mandel et al., 1993).
Está relacionada con cambios estructurales de las proteínas de diferentes virus, para un exitoso proceso infectivo a las células:
En el caso del virus HIV-1, los cambios conformacionales en la envoltura lipídica del HIV (Env), dependen de la acción coordinada de un sistema tripartita en el cual la PDI trabaja concertadamente con el receptor y correceptor, CD4 y CXCR4 respectivamente, al parecer inmersos en microdominios lipídicos “rafts” que sirven de plataforma y facilitan el proceso de entrada del virus (Markovic et al., 2003, Ou y Silver., 2006).
Virus como el Sindbis, el baculovirus y vaccinia también dependen de la actividad disulfuro isomerasa para la entrada a la célula, aunque el mecanismo catalítico del intercambio tiol/disulfuro en éstos no ha sido definido (Abell BA, et al.. 1993; Markovic et al., 1998; Locker et al., 1999).
En células MA104 y en las Caco-2, nosotros localizamos a PDI en los microdominios “rafts” de la membrana celular, formando complejos proteicos con los receptores de rotavirus, las proteínas Hsc70 e integrina v3. HSC70 ya ha sido reportada en los microdominios raft en células MA104 (López S 2006, Isa P. 2004), sin embargo la característica del complejo proteíco PDI-Hsc70 y av3 es la primera vez que se reporta.
El rotavirus en su proceso de ensamblaje hace uso de las “chaperonas” residentes en el retículo endoplasmatico para plegar sus proteínas estructurales, la conformación de la proteína VP7 es altamente susceptible a la inactivación de la proteína disulfuro isomerasa, ya que este proceso inhibe la formación de los puentes disulfuro nativos de VP7 y por ende se inhibe el ensamblaje del virus, observándose este efecto como acumulación de DLPs en el lumen del reticulo endoplasmatico (Svensson et al., 1994; Mirazimi et al. 1998, Delmas, et al., 2004; Maruri-Avidal, et al., 2008).
Dado que ese remodelaje de las proteínas estructurales a nivel de grupos tioles es importante para el ensamblaje el virus:
En este trabajo quisimos explorar si para la interacción con los receptores celulares, en el proceso de internalización, este proceso redox también está implicado.
Los resultados mostraron que la inhibición de la actividad disulfuro isomerasa de la superfi cie celular mediante DTNB y bacitracina disminuye la infección rotaviral en un modelo dosis dependiente y con un perfil saturable, sugiriendo que el rotavirus requiere de intercambios tiol-disulfuro en el transcurso de la infección celular.
El diseño experimental realizado indicó que la interacción del rotavirus mediada por grupos tioles transcurre en los eventos iniciales del proceso infeccioso en una etapa de pos-adherencia, ya que ni el DTNB ni la bacitracina impidieron la unión del virus a las células.
Además, no hubo variación de los porcentajes de infección comparados con células infectadas sin tratamiento, cuando estos reactivos se adicionaron en la etapa de post-entrada.
Más aun, los inhibidores no afectan el estado redox de las TLPs de RRV ya que al adicionarse a éstas, el porcentaje de infección celular fue comparable al de las partículas sin tratar.
Este resultado y el hecho que las TLPs de RRV no reaccionaran con el reactivo EZ-link Maleimide PEO2-Biotin indican la naturaleza oxidada de las proteinas virales. Cuando las células se incubaron con DTNB y luego éste fue removido por lavados antes del reto con el virus, se observó que la infección celular alcanzó porcentajes
La inhibición de la infección rotaviral, en células MA104:
En presencia de anticuerpos anti-PDI fue dosis- dependiente y como en el caso del DTNB mostró un perfi l saturable, con una inhibición del 90% a 10 μg/ml de anticuerpo anti-PDI. La inhibición por bloqueo de la PDI con anticuerpos, apoya los resultados obtenidos con los inhibidores químicos y sugieren que la PDI de la superfi cie celular interacciona con el rotavirus durante el proceso de entrada a la célula, en una etapa posterior a la unión del virus a la célula.
La inhibición de la infección utilizando anticuerpos no descarta la actividad del tipo chaperona de la PDI sobre el rotavirus, porque al adicionar el anticuerpo anti-PDI se puede estar inactivando esta función; en cambio cuando se inhibe la infección con DTNB o la bacitracina se está alterando es la función de intercambio tiol-disulfuro de la PDI.
Cuando se bloquea la PDI celular con anticuerpos la inhibición es hasta un 90%; mientras que bacitracina y DTNB inhiben la infección en un 70-75%. Esta diferencia en la inhibición se podría explicar por que el anticuerpo y los reactivos inhiben funciones diferentes de la PDI.
Este comportamiento es diferente al observado en el modelo de difteria y VIH, donde el 100% de inhibición de la infección se alcanzó con el aumento de la concentración de DTNB, mientras que con los anticuerpos anti-PDI la inhibición fue menor (Mandel et al., 1993; Ryser et al., 1991; 1994).
Una interacción directa entre las partículas rotavirales y PDI fue evidenciada en un sistema libre de células, utilizando PDI soluble:
Analizada mediante western blot y ELISA. Esa interacción también fue analizada en un sistema celular en el cual se marcó con biotina la PDI exterior y se bloquearon las cisteínas internas de la célula con NEM, luego de adherido el virus RRV a la célula; se logró detectar la presencia de PDI marcada con biotina y al menos VP4 en la fracción enriquecida en membranas, correspondiente a los microdominios lípidicos rafts, implicando la conformación de complejos proteícos en los que se incluye PDI-rotavirus en el proceso de entrada.
Para determinar que dominios de las proteínas viales interactúan con PDI, se seleccionaron secuencias peptídicas con cisteínas, que en el caso de VP7, éstas son totalmente conservadas en las diferentes cepas rotavirales consultadas que incluyeron cepas humanas, de porcino, de bovino y de simio.
Los péptidos sintéticos de las regiones 200-219 de la proteína VP8, 189-210 y 243-264 de la proteína VP7 de la cepa RRV, están involucrados en el proceso de entrada del rotavirus a la célula ya que tanto los péptidos incubados con las células, como los anticuerpos dirigidos contra éstos inhiben la infección rotaviral.
Al incubar las células con los péptidos modificados (Cys por Ser), se requiere mayor concentración de estos últimos para alcanzar el 50% de inhibición de la infección rotaviral, indicando una posible disminución en la afi nidad, probablemente mediada por las proteínas de actividad disulfuro isomerasa de la superficie celular.
Los estudios publicados sobre la unión de PDI a péptidos indican interacciones tanto con el sitio catalítico redox como por su región “chaperona”:
Entre ellos, la interacción de PDI con la cadena polipeptídica de 28 residuos, aminoterminal de la nucleasa del staphylococus, y también la somatostatina (Klappa, et al 1997; Quan et al., 1995; Xiaou R 2005). En estos casos, la afi nidad de unión péptido-PDI aumenta cuando se incrementa la longitud del péptido, y para péptidos de igual longitud, aquellos que contienen cisteínas se unen 4 a 8 veces mas fuertemente a PDI (Morjana y Gilbert, 1991).
Los resultados obtenidos con los péptidos sintéticos de las proteínas VP4 y VP7 son compatibles con lo reportado por Klappa et al, (1997 y Morjana y Gilbert (1991). En nuestro caso, explica la menor afi nidad mostrada por el P9 sin cisteína, en el ensayo de inhibición de la infección rotaviral, en el cual para alcanzar el 50% de inhibición se necesitó hasta 0.90 mM de concentración del péptido.
En cambio, esta misma inhibición se alcanzó con 0.34 mM de concentración del péptido original con cisteína, P8 de VP4 (Fig. 8B). Igual comportamiento fue encontrado con las secuencias peptídicas de VP7, en las que los péptidos originales con cisteínas presentaron afinidades mayores a las obtenidas con los péptidos modificados con serina. En este caso, para el péptido P2 la afinidad fue 4.5 veces mayor que para el péptido P3.
Según estos resultados de afinidad:
Relacionados con las concentraciones inhibitorias al 50%, sugieren que la secuencia de VP7 243-264 presenta una mayor afinidad a la célula con 0.18 mM, le sigue la secuencia de VP4 200-219 con 0.34 mM, y luego la secuencia de VP7 189-210 con 1.09 mM.
Todos los péptidos modifi cados (cambio de Cys por Ser) presentaron mayor valor de concentración inhibitora al 50% al compararlas con el péptido original. Se han descrito proteínas que no son miembros de la familia PDI que muestran algún grado de actividad disulfuro isomerasa, algunas de ellas son las integrinas, la proteína espliceosomal humana y la fibronectina.
Las integrinas tienen 9 repeticiones CXXC en cada subunidad, en los dominios ricos en cisteínas; la actividad tiol-disulfuro ha sido medida en experimentos libres de células, en procesos de renaturación de enzimas como la Ribunucleasa A; ensayos de inhibición de la actividad se han obtenido al incubar estas proteínas con bacitracina (O’Neill et al., 2000; Reuter, K. 1999; Langenbach et al., 1999; Weston et al., 2001).
En este trabajo no se puede descartar la probabilidad de que el dominio 3, pueda también infl uir en este proceso redox en rotavirus ya que justamente, el péptido rotaviral CPN (161-169) de VP7 es el ligando de esta integrina en rotavirus. Sin embargo, el cambio de la Cys por Ser en este trabajo indicó que no afecta el porcentaje inhibitorio de la infección que ejerce el péptido CPN.
Se ha reportado que la proteína transmembranal TM-SU del virus de la leucemia murina está implicada en la actividad de fusión membranal:
Para cual exhibe actividad de isomerasa que actúa sobre sus propios enlaces tiol-disulfuro por poseer es proteína viral en su secuencia el motivo catalítico CXXC (Wallin et al., 2004), motivo que está ausente en el caso de las proteínas rotavirales.
No se ha determinado si el rotavirus utiliza todas las moléculas candidatas a receptores, hasta ahora publicadas, o solo alguna de ellas, dependiendo de la línea celular y de la cepa de rotavirus.
Probablemente algunos rotavirus inicialmente utilizan las fracciones de ácido siálico y otros glicoconjugados, por el dominio galectina de la proteína VP8 (Dormitzer et al., 2002).
Las interacciones secuenciales con las proteínas de la célula, probablemente dependen de qué receptores tiene la célula. Quizá diferentes cepas utilizan una o varias proteínas dependiendo de la disponibilidad en cada célula.
Quizá también utilicen diferentes dominios de las proteínas virales, para producir los cambios conformacionales que el virus necesita, uniéndose en diferente momento con la misma proteína celular. Así, otras regiones expuestas por las que continuaría la interaccion podría involucrar la parte carboxi Terminal de VP8 analizada en este trabajo, la región de 200 a 219, que podría ser de interacción con PDI.
Igualmente está las interacciones con diferentes integrinas:
Entre éstas, la integrina 12 con el motivo DGE de VP5 y los diferentes dominios de VP7 con las integrinas 14, v3, x2 y Hsc70 (Graham et al., 2003, 2005; Guerrero et al., 2000, 2002; Zárate et at., 2003, 2004; Coulson et al., 1997; López et al. 2006).
Una de las interacciones de pos-adherencia es la región carboxi-terminal de VP5 con Hsc70 en un paso que es posterior a la temprana permeabilización de la membrana celular; además de la que involucra a VP6 con Hsc70 (Pérez-Vargas et al., 2006; Gualtero et al 2007). En todos estos pasos PDI podría estar involucrada, dado que el virus necesita la actividad tiol-redox para tener los cambios conformacionales indispensables para la unión sucesiva con las proteínas de la célula, durante su internalización celular.
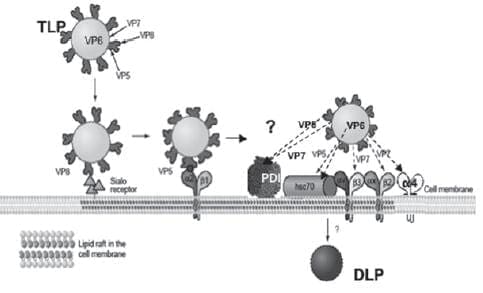
FIGURA 15. Mecanismo propuesto para la entrada de rotavirus, con adición de la participación de la PDI en un evento de post-unión pero antes de la internalización. Imagen modificada y adaptada de: López y Arias, 2004, y otras fuentes a saber: Graham et al., 2003, 2005; Superti et al., 1991; Isa, P et al., 1997; Rolsma, et al., 1998; Guo et al., 1999; Jolly et al 2000; Fiore et al 1991; Fuentes et al 1995; Dormitzer et al., 2002, Guerrero et al., 2000, 2002; Zarate et al., 2003, 2004; Coulson et al., 1997; Gualtero et at., 2007.
La incorporación de PDI al mecanismo propuesto de entrada del rotavirus se describe en la figura 15.
Lecturas Recomendadas
- 1. Abell BA, Brown DT. 1993. Sindbis virus membrane fusión is mediated by reduction of glycoprotein disulfi de bridges at the cell surface. J Virol 67:5496-5501
- 2. Altenburg, B. C., D. Y. Graham, and M. K. Estes 1980. Ultrastructural study of rotavirus replication in cultured cells. J Gen Virol. 46:75-85.
- 3. Angel, J., Franco, M.A. and Greenberg, H.B. 2007. Rotavirus vaccines: recent developments and future considerations. NATURE REVIEWS. MICROBIOLOGY. 5: 529-39
- 4. Arias, C.F, Guerrero, CA, Méndez, E, Zárate, S, Isa, P, Espinosa, R, Romero, P López, S. 2000. Early events of rotavirus infection: the search for the receptor(s). In Gastroenteritis Viruses. MK Estes and U Desselberger ed. John Wiley and Sons Ltd. New York.
- 5. Arias, C. F., P. Romero, V. Alvarez, and S. López 1996. Trypsin activation pathway of rotavirus infectivity. J Virol. 70:5832-5839.
- 6. Bass, D. M., M. R. Baylor, C. Chen, E. M. Mackow, M. Bremont, and H. B .
- 7. Ciarlet, M. 2002b. VLA-2 (alfa2beta1) integrin promotes rotavirus entry into cells but is not necessary for rotavirus attachment. J. Virol. 76, 1109-1123
- 8. Ciarlet, M. 2002a Initial interaction of rotavirus strains with N-acetylneuraminic (sialic) acid residues on the cell surface correlates with VP4 genotype, not species of origin. J. Virol. 76, 4087-4095
- 9. Conner,M., S., Blutt. 2007 Rotavirus: to the gut and beyond! Gastrointestinal infections Current Opinion in Gastroenterology. 23(1):39-43
- 10. Coulson, B. S., S. H. Londrigan, D. J. Lee 1997. Rotavirus contains integrin ligand sequences and a disintegrin-like domain implicated in virus entry into cells. Proc. Natl. Acad. Sci. USA.94:5389-5394.
Referencias
- 11. Cuadras M., Méndez E., Arias CF., López S. 1998. A new cysteine in rotavirus VP4 participates in the formation of an alternate disulfi de bond. Journal of General Virology. 79, 2673-2677.
- 12. Delorme C, Brüssow H, Sidoti J, Roche N, Karlsson KA, Neeser JR, Teneberg S. 2001. Glycosphingolipid binding specifi cities of rotavirus: identifi cation of a sialic acid-binding epitope. J Virol, 75:2276-2287.
- 13. Dennehy P.H. 2007. Rotavirus vaccines-An update Vaccine 25 (3137-3141 3141.
- 14. Dormitzer, P.R. et al. 2002 Specifi city and affi nity of sialic acid binding by the rhesus rotavirus VP8* core. J. Virol. 76, 10512-10517.
- 15. Essex, D, Chan K, Swiatkowska M. 1995. Localization of PDI to the external surface of the platelet plasma membrane. Blood, 86(6):2168-2173.
- 16. Estes M. K., Graham, DY, y Manso, B.B. 1981. Proteolytic enhancement of rotavirus infectivity: molecular mechanisms. J. Virol., 39, 879-888.
- 17. Estes, M. K. (1996). Rotaviruses and their replication. In “Virology” (N. Fields, D. M. Knipe, and P. M. Howley, Eds.), 3rd ed., pp. 1625-1655. Raven Press, New York.
- 18. Fiore, L. 1991. The VP8 fragment of VP4 is the Rhesus rotavirus hemagglutinin. Virology 181: 553-563.
- 19. Freedman, R.B., P. Klappa. 1999. In Bukau, B. (Ed). Protein Disulfi de Isomerase: A catalyst of thiol:disulfi de Interchange and Associated Protein Folding. Harwood Academic Press, London, UK, pp. 437-459.
- 20. Graham, D. Y. and M. K. Estes. 1988. Viral infections of the intestine, p.566-578. In G. Gitnick (ed.), Gastroenterology. Medical Examination Publishing Company, New Hyde Park, N.Y.
- 21. Graham, K.L. 2003. Integrin-using rotaviruses bind alpha2- 1 integrin alpha2 I domain via VP4 DGE sequence and recognize alphaX2 and alphaV3 by using VP7 during cel l en try. J. Virol. 77, 9969-9978
Bibliografías
- 22. Gajardo, R., P. Vende, D. Poncet, and J. Cohen 1997. Two proline residues are essential in the calcium-binding activity of rotavirus VP7 outer capsid protein. J. Virol. 71:2211- 2216.
- 23. Greenberg 1992. Liposome-mediated transfection of intact viral particles reveals that plasma membrane penetration determines permissivity of tissue culture cells to rotavirus. J Clin Invest. 90:2313-20.
- 24. Guerrero, C. A., E.Méndez, S. Zárate, P. Isa, S. López, and C. F. Arias. 2000. Integrin alpha(v)(3) mediates rotavirus cell entry. Proc. Natl. Acad. Sci. USA 97:14644-14649.
- 25. Guerrero, C.A. 2000. Biochemical characterization of rotavirus receptors in MA104 cells. J. Virol. 74, 9362-9371
- 26. Guerrero, C. A. 2000. Identifi cación y caracterización del receptor de Rotavirus independiente de ácidos siálicos. Tesis doctoral, Doctorado en Ciencias Bioquímicas. Universidad Autónoma de México
- 27. Guerrero, C. A., D. Bouyssounade, S. Zarate, R. Espinosa, P. Romero, E. Mendez, S. López, and C. F. Arias. 2002. The heat shock cognate protein 70 is involved in rotavirus cell entry J Virol. 76:4096-4102.
- 28. Hewish, M. J., Y. Takada, B. S. Coulson 2000. Integrins X2 and X41 can mediate SA11 rotavirus attachment and entry into cells. J Virol. 74:228-236.
- 29. Jayaram H, Estes MK, Prasad BV. (2004). Emerging themes in rotavirus cell entry, genome organization, transcription and replication. Virus Res. 101(1): 67-81.
- 30. Jiang, X. M., Fitzgerald, M., Grant, C. M., and Hogg, P. J. (1999) J. Biol. Chem.274, 2416-2423.
- 31. Jimena Pérez-Vargas, Pedro Romero, Susana Lo´pez, and Carlos F. Arias*2006. The Peptide-Binding and ATPase Domains of Recombinant Hsc70 Are Required To Interact with Rotavirus and Reduce Its Infectivity.JOURNAL OF VIROLOGY, Apr., p. 3322-3331 Vol. 80, No. 7.
Fuentes
- 32. Jolly, C.L. et al. (2000) Rotavirus infection of MA104 cells is inhibited by Ricinus lectin and separately expressed single binding domains. Virology 275, 89-97
- 33. Kapikian, A. Z., R. M. Chanock. 2000. Rotaviruses. In B. N. Fields, D. N. Knipe, P. M. Howley, R. M. Chanock, J. L. Melnick, T. P. Monath, B. Roizman, S. E. Straus (eds), Virology, vol. 2. Raven Press, New Cork, p. 1787-1833
- 34. Kluwer Academic Publishers, Yoshimori, T., Semba, T., Takemoto, H., Akagi, S., Yamamoto, A., and Tashiro, Y. (1990) J. Biol. Chem. 265, 15984-15990.
- 35. Langenbach KJ, Sottile J. 1999. Identifi cation of proteindisulfide isomerase activity in fibronectin. J Biol Chem 274:7032-7038.
- 36. Locker JK, Griffi ths G. 1999. An unconventional role for cytoplasmic disulfi de bonds in vaccinia virus proteins. J Cell Biol.144:267-279
- 37. López, S., C. F. Arias. 2004. Multistep entry of rotavirus into cells: a Versaillesque dance. TRENDS in Microbiology 12: 271-278
- 38. López, S., Espinosa, R., Isa, P., Zárate, S., Méndez, E., Arias, C.F. 2000. Characterization of a Monoclonal Antibody Directed to the Surface of MA104 Cells that Blocks the Infectivity of Rotaviruses. Virology 273:160-168
- 39. López, T., López S., Arias CF. 2006. Heat shock enhances the susceptibility of BHK cells to rotavirus infection through the facilitation of entry and post-entry virus replication steps. Virus Research, 121 (1):74-83
- 40. López, S., Arias, C. F., Bell, J. R., Strauss, J. H., and Espejo, R. T. (1985). Primary structure of the cleavage site associated with trypsin enhancement of rotavirus SA11 infectivity. Virology 144, 11-19.
- 41. Macer DR, Koch GL. 1988. Identifi cation of a set of calciumbinding proteins in reticuloplasm, the luminal content of the endoplasmic reticulum. J Cell Sci 91:61-70.
Referencias Bibliográficas
- 42. Mahmoud S. Shah Rabadi, Lorne A. Babiuk, Patrick W. K. Lee, 1987.Further analysis of the role of calcium in rotavirus morphogenesis. Virology, Volume 158, Issue 1, May, Pages 103-111
- 43. Mandel R, Ryser HJ, Ghani F, Wu M, Peak D. 1993. Inhibition of a reductive function of the plasma membrane by bacitracin and antibodies against protein disulfi de-isomerase. Proc Natl Acad Sci USA 90:4112-4116
- 44. Markovic I, Pulyaeva H, Sokoloff A, Chernomordik LV. 1998. Membrane fusion mediated by baculovirus gp64 involves assembly of stable gp64 trimers into multiprotein aggregates. J Cell Biol.;143:1155-1166
- 45. Markovic, I., T. S. Stantchev, K. H. Fields, L. J. Tiffany, M. Tomi, C. D. Weiss, C.C. Broder, K. Strebel, K. A. Clouse. 2003. Thiol/disulfi de exchange is a pre-requisite for CXCR4- tropic HIV-1 envelope mediated T-cell fusion during viral entry. Blood. First Edition Paper (05):1390
- 46. Mathieu, M., I. Petitpas, J. Navaza, J. Lepault, E. Kohli, P. Pothier, B. V. Prasad, J. Cohen, and F. A. Rey 2001. Atomic structure of the major capsid protein of rotavirus: implications for the architecture of the virion. Embo J. 20:1485-1497.
- 47. Matthijnssens, J. et al. Full genomic analysis of human rotavirus strain B4106 and lapine rotavirus strain 30/96 provides evidence for interspecies transmission. J. Virol. 80, 3801-3810 (2006).
- 48. Mosman, T. 1983. Rapid colorimetric assay for cellular growth and survival: application of proliferation and cytotoxicity assay. J Immunol Methods. 65:55-63
- 49. Mossel, E.C. and R.F. Ramig. 2003. A lymphatic mechanism of rotavirus extraintestinal spread in the neonatal mouse. J. Virol. 77, 12352-12356
- 50. Méndez, E., López, S., Cuadras, M. A., Romero, P., and Arias, C. F. 1999. Entry of rotaviruses is a multistep process. Virology 263: 450-459.
Fuentes Bibliográficas
- 51. M. Fenaux, M. A. Cuadras, N. Feng, M. Jaimes, and H. B. Greenberg* Extraintestinal Spread and Replication of a Homologous EC Rotavirus Strain and a Heterologous Rhesus Rotavirus in BALB/c Mice. J. Virol. 2006, Vol. 80, No. 11, p. 5219-5232
- 52. O’Neill S, Robinson A, Deering A, Ryan M, Fitzgerald DJ, Moran N. 2000. The platelet integrin aII3 has an endogenous thiol isomerase activity. J Biol Chem 275:36984-36990.
- 53. Ou W, Silver J. 2006. Role of protein disulfi de isomerase and other thiol-reactive proteins in HIV-1 envelope proteinmediated fusion. Virology 350: 406-417
- 54. Parashar UD, Gibson CJ, Bresse JS, Glass RI. Rotavirus and severe childhood diarrhea. Emerg Infect Dis 2006;12(2):304-6.
- 55. Patton, J. T., J. Hua, and E. A. Mansell 1993. Location of intrachain disulfi de bonds in the VP5* and VP8* trypsin cleavage fragments of the rhesus rotavirus spike protein VP4. J Virol. 67:4848-4855.
- 56. Pavel I, Realpe M., Romero P, López S. , Arias CF. 2004. Rotavirus RRV associates with lipid membrane microdomains during cell entry. Virology, 322, (2): 370-38.
- 57. Pesavento, J.B., Estes, M.K., Prasad, B.V.V., 2003. Structural organization of the genome in rotavirus. In: Desselberger, U., Gray, J. (Eds.), Viral Gastroenteritis, fi rst ed., vol. 9. Elsevier Science, Amsterdam, pp. 115-128
- 58. Prasad, B. V., G. J. Wang, J. P. Clerx, and W. Chiu 1988. Three-dimensional structure of rotavirus. J Mol Biol. 199:269- 275
- 59. Ramig RF. 2004 Pathogenesis of intestinal and systemic rotavirus infection. J Virol. 78(19):10213-20.
- 60. Reuter K, Nottrott S, Fabrizio P, Luhrmann R, Ficner R. 1999. Identifi cation, characterization, and crystal structure analysis of the human spliceosomal U5 snRNP-specifi c 15 kDa protein. J Mol Biol 294:515-525.
Otras Lecturas Recomendadas
- 61. Ryser, H. J. P., Mandel, R., Gallina, A., and Rivera, A. (1997) in Propyl 4-Hydroxylase, Protein Disulfi de Isomerase and Other Structurally Related pProteins (Guzman, N. A., ed) pp. 425-446, Marcel Dekker, Inc., New York
- 62. Ryser, H. J. P., Mandel, R., and Rivera, A. (1999) in Plasma Membrane Redox Systems and Their Role in Biological Stress and Disease (Asard, H., Berczi, A., and Caubergs, R. J., eds) pp. 229-307.
- 63. Ryser HJ, Levy EM, Mandel R, DiSciullo GJ. 1994. Inhibition of human immunodefi ciency virus infection by agents that interfere with thiol-disulfi de interchange upon virus receptor interaction. Proc Natl Acad Sci U S A. 91:4559-4563
- 64. Schwaller M, Wilkinson B, Gilbert HF. 2003. Reduction-reoxidation cycles contribute to catalysis of disulfi de isomerization by protein-disulfi de isomerase. J Biol Chem. 278(9):7154- 9.
- 65. Superti F, G. Donelli 1991. Gangliosides as binding sites in SA-11 rotavirus infection of LLC-MK2 cells. J Gen Virol. Oct;72 ( Pt 10):2467-74
- 66. Svensson, L., P. R. Dormitzer, B. C. von, L. Maunula, and H. B. Greenberg 1994. Intracellular manipulation of disulfi de bond formation in rotavirus proteins during assembly. J. Virol. 68:5204-5215.
- 67. Tager M, Kroning H, Thiel U, Ansorge S. 1997. Membranebound proteindisulfi de isomerase (PDI) is involved in regulation of surface expression of thiols and drug sensitivity of B-CLL cells. Exp Hematol 25:601-607.
- 68. Trask SD, Dormitzer PR. 2006 Assembly of highly infectious rotavirus particles recoated with recombinant outer capsid proteins. J Virol 80: 11293-11304
- 69. Terada, K., Manchikalapudi, P., Noiva, R., Jauregui, H. O., Stockert, R. J., and Schilsky, M. L. (1995) J. Biol. Chem. 270, 20410-20416.
Otras Referencias
- 70. Turano, C., S. Coppari, F. Altieri, A. Ferraro. 2002. Proteins of the PDI Family: Unpredicted Non-ER Locations and Functions. J. Cell. Phys. 193:154-163
- 71. Wallin M., Ekstro¨m M., Garoff H. 2004. Isomerization of the intersubunit disulphide-bond in Env controls retrovirus fusion. The EMBO Journal 23, 54-65
- 72. Weston, BS., Wahab, N., Roberts, T., Mason, R. 2001. Bacitracin inhibits fi bronectin matrix assembly by mesangial cells in high glucose. Kidney International, Vol. 60: 1756-1764
- 73. Yaeger, M., M. Anderson. 2003. Rotavirus Still Impact Bottom Line. TGE, National Hog Farmer p. 3-5
- 74. Zai, A., Rudd, M. A., Scribner, A. W., and Loscalzo, J. (1999) J. Clin. Invest. 103, 393-399.
- 75. Zárate, S., M. A. Cuadras, R. Espinosa, P. Romero, K. O. Juárez, M. Camacho-Nuez, C. F. Arias, S. López. 2003. Interaction of Rotaviruses with Hsc70 during Cell Entry Is Mediated by VP5. J Virol. p. Vol. 77(13): 7254-7260
- 76. Zárate, S., R. Espinosa, P. Romero, E. Méndez, C. F. Arias, S. López 2000. The VP5 domain of VP4 can mediate attachment of rotaviruses to cells. J Virol. 74:593-599
- 77. Zárate, S., R. Espinosa., P. Romero, C. A. Guerrero., C. F. Arias, S. López. 2000. Integrin alpha 2 1 mediates the cell attachment of the rotavirus neuraminidase-resistant variant nar3. Virology. Vol 278 p 50 – 54
- 78. Zárate S, Cuadras MA, Espinosa P, Romero P, Juarez KO, Camacho-Nuez M, Arias CF, Lopez S. 2003. Interaction of rotaviruses with HSC70 during cell entry is mediated by VP5. J Virol 77: 7254-7260.
- 79. Zárate S, Romero P, Espinosa R, Arias CF, Lopez S . 2004. VP7 mediates the interaction of rotaviruses with integrin v3 through a novel integrin-binding site. J Virol 78: 10839- 10847.
CLIC AQUÍ Y DÉJANOS TU COMENTARIO